标签:ISE 设置 plt direct tga 源码 ima problem port
园子里关于遗传算法的教案不少,但基于geatpy框架的并未多见,故分享此文以作参考,还望广大园友多多指教!
Geatpy出自三所名校联合团队之手,是遗传算法领域的权威框架(python),其效率之高、应用领域之广远胜诸多第三方工具,此处不作赘述,直接上链接:
官网:http://www.geatpy.com/start
源码:https://github.com/geatpy-dev/geatpy/tree/master/geatpy
使用Geatpy需要安装geatpy模块(pip install geatpy),linux下如果装完后import时出现报错,可以下载我帖尾链接里的wheel文件进行安装。
言归正传,根据经典的遗传算法流程,无外乎这几个步骤:种群初始化 ->(适应度评价 -> 遴选 -> 交叉 -> 变异)<- 循环进化直至终止条件达标。
当然,有关遗传算法的原理和过程不做深讨,本文旨在剖析遗传算法在阵曲线寻优上的高效应用,我写了一个简单示例来帮助大家更好的理解,代码如下:
# -*- coding: utf-8 -*- """punishing.py - 罚函数demo""" import numpy as np def punishing(LegV, FitnV): FitnV[np.where(LegV == 0)[0]] = 0 return FitnV
# -*- coding: utf-8 -*- """ aimfc.py即目标函数,本例通过输入每一代的染色体,由自定义评价函数计算与目标曲线的面积差,作为目标函数值ObjV来输出 """ import numpy as np def MakeObjCurve(width): ‘‘‘ 创建目标曲线,此处定义为一组正弦波拼接序列 ‘‘‘ n1 = width//3 n2 = width - 2*n1 x1=np.cos(np.arange(0,n1)) * 1 x2=np.cos(np.arange(0,n1)) * 4 x3=np.cos(np.arange(0,n2)) * 2 ObjCurve=np.hstack((x1,x2,x3)) return ObjCurve def CalScore(chrom): ‘‘‘ 返回染色体与目标曲线之间的面积的倒数作为评分值 ‘‘‘ objCurve = MakeObjCurve(len(chrom)) area = chrom - objCurve area *= 10**5 #调整系数确保分值不受小数项干扰 score = 1 / np.dot(area, area) #计算差值的平方和以简化求面积过程 return score def myEvaFunc(chroms): ‘‘‘ 自定义评价函数,以评分值作为目标函数值 ‘‘‘ scores = [] for chrom in chroms: score = CalScore(chrom) scores.append(score) scores = np.array([scores]).T return scores def aimfuc(Phen, LegV): ObjV = myEvaFunc(Phen) exIdx = np.argmin(ObjV[:, 0]) # 惩罚方法2: 标记非可行解在可行性列向量中对应的值为0,并编写punishing罚函数来修改非可行解的适应度。 # 也可以不写punishing,因为Geatpy内置的算法模板及内核已经对LegV标记为0的个体的适应度作出了修改。 # 使用punishing罚函数实质上是对非可行解个体的适应度作进一步的修改 LegV[exIdx] = 0 # 对非可行解作出标记,使其在可行性列向量中对应的值为0,此处标记的是得分最小项 return [ObjV, LegV]
# -*- coding: utf-8 -*- """ main.py即主函数,本例仅用于演示“已知曲线寻优”的过程 """ import numpy as np import geatpy as ga import time import matplotlib.pyplot as plt def search_objects(directory): directory=os.path.normpath(directory) #规格化,防止分隔符造成的差异 if not os.path.isdir(directory): raise IOError("The directory ‘"+"‘ doesn‘t exist!") objects={} for curdir,substrs,files in os.walk(directory): for jpeg in (file for file in files if file.endswith(‘.csv‘)): path=os.path.join(curdir,jpeg) label=path.split(os.path.sep)[-2] if label not in objects: objects[label]=[] objects[label].append(path) return objects def sga_mps_real_templet(AIM_M, AIM_F, PUN_M, PUN_F, FieldDRs, problem, maxormin, MAXGEN, NIND, SUBPOP, GGAP, selectStyle, recombinStyle, recopt, pm, distribute, drawing = 1): """ 基于多种群独立进化单目标编程模板(实值编码),各种群独立将父子两代合并进行选择,采取精英保留机制 """
#==========================初始化配置=========================== GGAP = 0.5 # 因为父子合并后选择,因此要将代沟设为0.5以维持种群规模 # 获取目标函数和罚函数 aimfuc = getattr(AIM_M, AIM_F) # 获得目标函数 if PUN_F is not None: punishing = getattr(PUN_M, PUN_F) # 获得罚函数 NVAR = FieldDRs[0].shape[1] # 得到控制变量的个数 # 定义全局进化记录器,初始值为nan pop_trace = (np.zeros((MAXGEN ,2)) * np.nan) pop_trace[:, 0] = 0 # 定义变量记录器,记录控制变量值,初始值为nan var_trace = (np.zeros((MAXGEN ,NVAR)) * np.nan) """=========================开始遗传算法进化=======================""" start_time = time.time() # 开始计时 # 对于各个网格分别进行进化,采用全局进化记录器记录最优值 for index in range(len(FieldDRs)): # 遍历各个子种群,各子种群独立进化,互相不竞争 FieldDR = FieldDRs[index] if problem == ‘R‘: Chrom = ga.crtrp(NIND, FieldDR) # 生成初始种群 elif problem == ‘I‘: Chrom = ga.crtip(NIND, FieldDR) LegV = np.ones((NIND, 1)) # 初始化种群的可行性列向量 [ObjV, LegV] = aimfuc(Chrom, LegV) # 求初始种群的目标函数值 repnum = 0 # 初始化重复个体数为0 ax = None # 存储上一帧图形 gen = 0 badCounter = 0 # 用于记录在“遗忘策略下”被忽略的代数 # 开始进化!! while gen < MAXGEN: if badCounter >= 10 * MAXGEN: # 若多花了10倍的迭代次数仍没有可行解出现,则跳出 break # 进行遗传算子,生成子代 SelCh = ga.recombin(recombinStyle, Chrom, recopt, SUBPOP) # 重组 if problem == ‘R‘: SelCh = ga.mutbga(SelCh,FieldDR, pm) # 变异 if repnum > Chrom.shape[0] * 0.01: # 当最优个体重复率高达1%时,进行一次高斯变异 SelCh = ga.mutgau(SelCh, FieldDR, pm) # 高斯变异 elif problem == ‘I‘: SelCh = ga.mutint(SelCh, FieldDR, pm) LegVSel = np.ones((SelCh.shape[0], 1)) # 初始化育种种群的可行性列向量 [ObjVSel, LegVSel] = aimfuc(SelCh, LegVSel) # 求育种种群的目标函数值 # 父子合并 Chrom = np.vstack([Chrom, SelCh]) ObjV = np.vstack([ObjV, ObjVSel]) LegV = np.vstack([LegV, LegVSel]) FitnV = ga.ranking(maxormin * ObjV, LegV, None, SUBPOP) # 适应度评价 if PUN_F is not None: FitnV = punishing(LegV, FitnV) # 调用惩罚函数 repnum = len(np.where(ObjV[np.argmax(FitnV)] == ObjV)[0]) # 计算最优个体重复数 # 记录进化过程 bestIdx = np.argmax(FitnV) if (LegV[bestIdx] != 0) and ((np.isnan(pop_trace[gen,1])) or ((maxormin == 1) & (pop_trace[gen,1] >= ObjV[bestIdx])) or ((maxormin == -1) & (pop_trace[gen,1] <= ObjV[bestIdx]))): feasible = np.where(LegV != 0)[0] # 排除非可行解 pop_trace[gen,0] += np.sum(ObjV[feasible]) / ObjV[feasible].shape[0] / len(FieldDRs) # 记录种群个体平均目标函数值 pop_trace[gen,1] = ObjV[bestIdx] # 记录当代目标函数的最优值 var_trace[gen,:] = Chrom[bestIdx, :] # 记录当代最优的控制变量值 # 绘制动态图 if drawing == 2: ax = ga.sgaplot(pop_trace[:,[1]],‘子种群‘+str(index+1)+‘各代种群最优个体目标函数值‘, False, ax, gen) badCounter = 0 # badCounter计数器清零 else: gen -= 1 # 忽略这一代(遗忘策略) badCounter += 1 if distribute == True: # 若要增强种群的分布性(可能会造成收敛慢) idx = np.argsort(ObjV[:, 0], 0) dis = np.diff(ObjV[idx,0]) / (np.max(ObjV[idx,0]) - np.min(ObjV[idx,0]) + 1)# 差分计算距离的修正偏移量 dis = np.hstack([dis, dis[-1]]) dis = dis + np.min(dis) # 修正偏移量+最小量=修正绝对量 FitnV[idx, 0] *= np.exp(dis) # 根据相邻距离修改适应度,突出相邻距离大的个体,以增加种群的多样性 [Chrom, ObjV, LegV] = ga.selecting(selectStyle, Chrom, FitnV, GGAP, SUBPOP, ObjV, LegV) # 选择 gen += 1 end_time = time.time() # 结束计时 times = end_time - start_time # 后处理进化记录器 delIdx = np.where(np.isnan(pop_trace))[0] pop_trace = np.delete(pop_trace, delIdx, 0) var_trace = np.delete(var_trace, delIdx, 0) if pop_trace.shape[0] == 0: raise RuntimeError(‘error: no feasible solution. (有效进化代数为0,没找到可行解。)‘) # 输出结果 if maxormin == 1: best_gen = np.argmin(pop_trace[:, 1]) # 记录最优种群是在哪一代 best_ObjV = np.min(pop_trace[:, 1]) elif maxormin == -1: best_gen = np.argmax(pop_trace[:, 1]) # 记录最优种群是在哪一代 best_ObjV = np.max(pop_trace[:, 1]) print(‘最优的目标函数值为:%s‘%(best_ObjV)) print(‘最优的控制变量值为:‘) for i in range(NVAR): print(var_trace[best_gen, i]) print(‘有效进化代数:%s‘%(pop_trace.shape[0])) print(‘最优的一代是第 %s 代‘%(best_gen + 1)) print(‘时间已过 %s 秒‘%(times)) # 绘图 if drawing != 0: ga.trcplot(pop_trace, [[‘种群个体平均目标函数值‘, ‘种群最优个体目标函数值‘]]) # 返回进化记录器、变量记录器以及执行时间 return [pop_trace, var_trace, times, best_gen] # 获取函数接口地址 AIM_M = __import__(‘aimfuc‘) PUN_M = __import__(‘punishing‘) POP_SIZE = 300 # 种群高度 CHROM_LENGTH = 20 # 染色体宽度 max_generation = 150 # 进化代数 chrom_bottom = -4 #染色体数值下限 chrom_top = 4 #染色体数值上限 # 变量设置 x = []; b = [] for i in range(CHROM_LENGTH): x.append([chrom_bottom, chrom_top]) # 自变量的范围 b.append([0, 0]) # 自变量是否包含下界 ranges=np.vstack(x).T # 生成自变量的范围矩阵 borders = np.vstack(b).T # 生成自变量的边界矩阵 precisions = [1]*CHROM_LENGTH # 在二进制/格雷码编码中代表自变量的编码精度,当控制变量是连续型时,根据crtfld参考资料,该变量只表示边界精度,故设置为一定的正数即可 # 生成网格化后的区域描述器集合 FieldDRs = [] for i in range(1): FieldDRs.append(ga.crtfld(ranges, borders, precisions)) # 调用编程模板(设置problem = ‘R‘处理实数型变量问题,详见该算法模板的源代码) [pop_trace, var_trace, times, best_gen] = sga_mps_real_templet(AIM_M, ‘aimfuc‘, PUN_M, ‘punishing‘,
FieldDRs, problem = ‘R‘, maxormin = -1, MAXGEN = max_generation, NIND = POP_SIZE, SUBPOP = 1, GGAP = 0.9, selectStyle = ‘tour‘, recombinStyle = ‘xovdprs‘, recopt = 0.9, pm = 0.3, distribute = True, drawing = 1) bstChrom = var_trace[best_gen] objCurve = AIM_M.MakeObjCurve(CHROM_LENGTH) plt.ion() fig = plt.figure(‘曲线寻优演示‘,facecolor=‘lightgray‘) ax1 = fig.add_subplot(2, 1, 1) ax2 = fig.add_subplot(2, 1, 2) ax1.set_title("Evaluation Map") ax1.grid(axis=‘y‘, linestyle=‘:‘) for i in range(max_generation): if i%5==0: ax1.plot(var_trace[i], ‘o-‘) ax2.cla() ax2.set_title("最优染色体[gen:%i]"%(i+1)) ax2.plot(var_trace[i], ‘o-‘, c=‘dodgerblue‘) plt.pause(0.001) ax2.cla() ax2.grid(axis=‘y‘, linestyle=‘:‘) ax2.plot(objCurve, ‘o-‘, c=‘orangered‘, label=‘目标曲线‘) ax2.plot(bstChrom, ‘o-‘, c=‘dodgerblue‘, label=‘最优染色体[gen:%i]‘%(best_gen+1)) plt.legend() plt.ioff() plt.show()
请注意,此处我已将模板函数单独放到主函数中以便大家更好的理解,返回值中增加了最优代数以便后续图例的显示。
本例采用的进化模板是sga_mps_real_templet,基于多种群进化单目标(实数值),用于实现寻找目标曲线。
目标曲线的定义函数在aimfc.py的MakeObjCurve函数中,本例为3段振幅不同的cos函数拼接而成的模拟曲线,宽度20。
种群初始值设置:种群高度300、染色体宽度20(与目标曲线宽度保持一致)、进化代数150、染色体数值上下限[-4,4](与目标曲线的上下限保持一致)。
接着我们开始寻优,通过CalScore函数计算每代种群的每条染色体与目标函数之间的差值,经过一定的系数转换得到评分值(差值越大,评分越低),以单目标(1列)形式输出,由geatpy的ranking函数来决定适应度评价,然后继续遴选、交叉、变异,如此循环往复,直至达到近似目标值时终止(本例设定为150代时终止),寻优过程如下图:
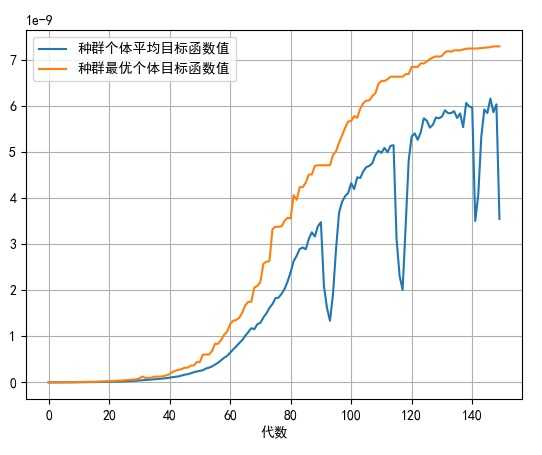
最优的目标函数值为:7.287567405118076e-09
有效进化代数:150
最优的一代是第 148 代
时间已过 1.3259999752044678 秒
可以看到从50代左右优化曲线开始显著上扬,直到130代左右逐渐平缓,并且耗时非常少,来看寻优结果图:
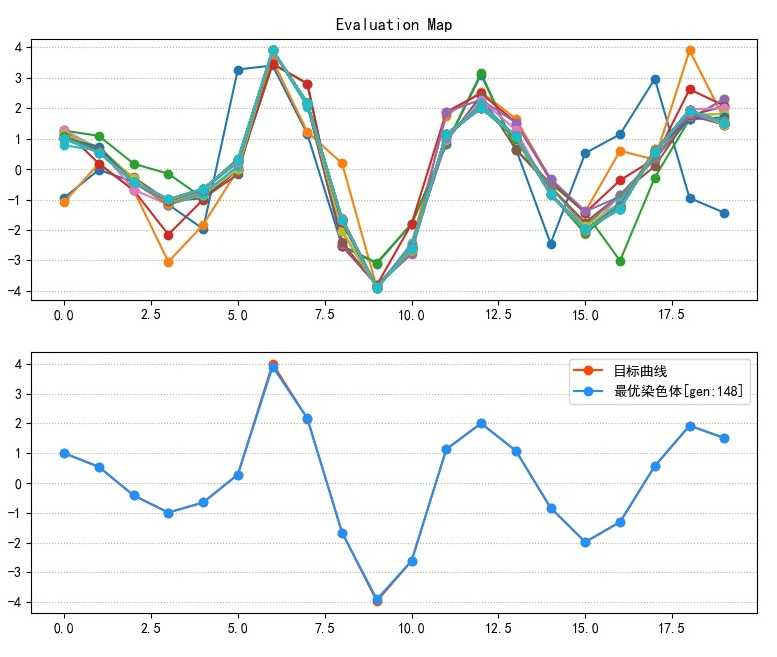
可以看到除了第6个点的数值有细微差异之外,其他点几乎都是吻合的,基本实现了目标曲线的寻求。
之所以抛出本例,最重要的一点在于geatpy遗传算法不仅能寻求已知的目标函数,还可以通过自定义的评分体系或第三方接口来参与实现寻优过程,只需将CalScore函数稍作改动即可,以上。
【wheel文件】: https://pan.baidu.com/s/1BwLq_m3Dd5RMqatvTYXrAw 提取码: vgkz
标签:ISE 设置 plt direct tga 源码 ima problem port
原文地址:https://www.cnblogs.com/dent-lu/p/10340709.html