标签:ola 变化 样本 过程 信号 can max test 使用
题目:Deep Protein Methylation Profiling by Combined Chemical and Immunoaffinity Approaches Reveals Novel PRMT1 Targets (结合层析法和免疫沉淀法的蛋白甲基化的深度检测技术发现了PRMT1新的靶标蛋白)
期刊名:Molecular & Cellular Proteomics (MCP)
发表日期:2019.11.1
IF: 4.828
DOI:10.1074/mcp.RA119.001625
作者及单位:
1、 Nicolas G. Hartel;Mork Family Department of Chemical Engineering and Materials Science
2、 Brandon Chew;Mork Family Department of Chemical Engineering and Materials Science
3、 Jian Qin;Center for Craniofacial Molecular Biology,Herman Ostrow School of Dentistry
4、 Jian Xu;Center for Craniofacial Molecular Biology,Herman Ostrow School of Dentistry
5、 Nicholas A. Graham(通讯作者);Mork Family Department of Chemical Engineering and Materials Science,Norris Comprehensive Cancer Center, University of Southern California, Los Angeles, California
一、 概述
蛋白质甲基化在很多生物过程中起着重要的作用,包括信号转导、代谢以及转录调控,但受制于技术的限制,目前还缺少在全局对所有蛋白的甲基化程度进行深度检测。蛋白甲基化含有多种修饰形式,主要发生在精氨酸和赖氨酸上,在赖氨酸上有三种修饰形式,分别为单甲基化(Kme1)、二甲基化(Kme2)和三甲基化(Kme3);精氨酸上的修饰形式也是三种,分别为单甲基化(monomethyl,MMA)、不对称二甲基化(asymmetric methyl, ADMA)和对称二甲基化(symmetric methyl, SDMA)。目前导致甲基化修饰的研究落后于其他修饰的主要原因在于缺乏成熟的甲基化肽段富集方法,常用的甲基化肽段检测方法主要是基于抗体对甲基化肽段的富集,但目前的抗体质量及特异性还有待提高;另一方面,当赖氨酸及精氨酸上发生甲基化修饰后,往往会影响胰蛋白酶的酶解,导致肽段的漏切,从而时甲基化肽段带电量增加,用强阳离子交换柱可以对甲基化肽段进行相对富集,这两种甲基化肽段的富集方法还没有进行过严谨的比较。
本篇研究比较了两种不同甲基化富集方法的效率,发现两者之间具有较好的正交性,可以互补,随后结合了两种富集方法对蛋白精氨酸甲基转移酶1(protein arginine methyltransferase 1, PRMT1)在细胞内形式甲基化功能的靶标做了全面的研究。
二、 实验方法及流程
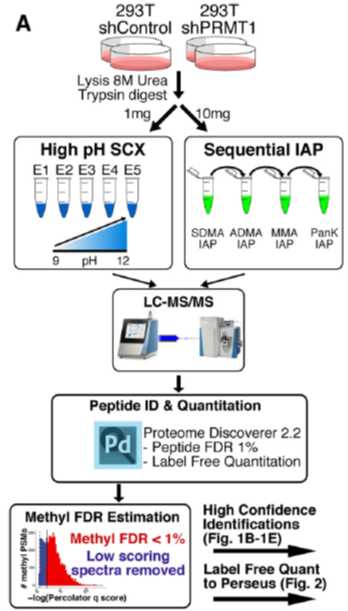
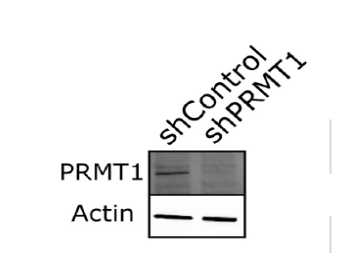
1、以293T细胞为材料,转入shPRMT1质粒来敲除PRMT1蛋白,并经过WB验证敲除效率;同时采用SCX法和免疫亲和法(IAP)对相同样品做处理,最终使用质谱进行检测,随后对结果进行定性和定量分析,每个样本都有两次生物重复,流程如图所示:
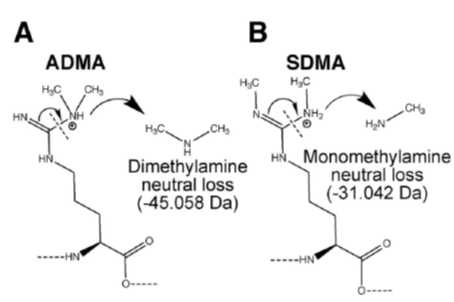
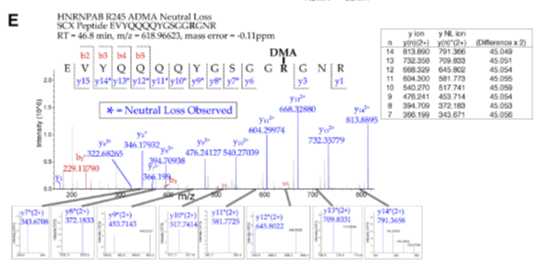
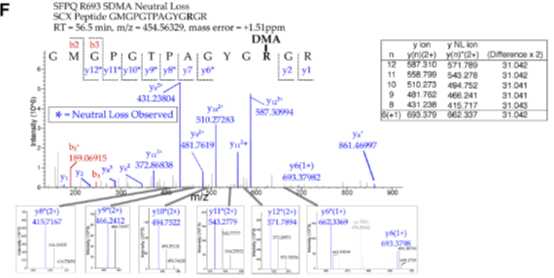
2、对于ADMA和SDMA两种甲基化修饰,采用中性丢失的搜库方法加以区分,原理及实际质谱图如下图所示:
在二级碎裂中,精氨酸上的胍基有概率在图示部分被打断,产生一个中性丢失集团,当精氨酸上的甲基化修饰类型不同时,掉下来的中性集团分子量也不同,通过这个特征可以将ADMA和SDMA有效的做出区分
三、 研究成果
1、 SCX和IAP可富集到不同的甲基化肽段
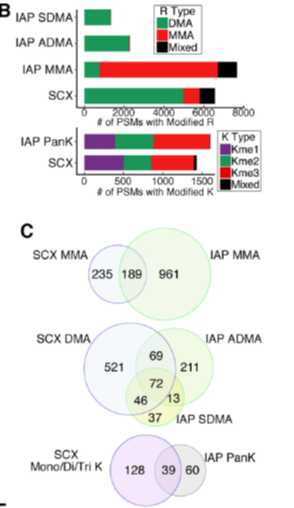
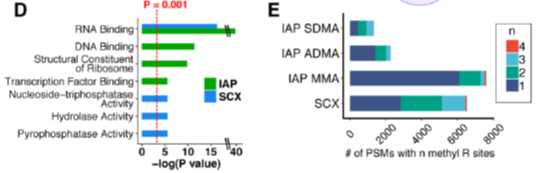
将结果只按照富集方法,而不考虑是否敲除PRMT1蛋白来分析时发现,两种富集方法鉴定到的不同类型甲基化肽段的交集均较低,图B展示了不同方法鉴定到甲基化肽段的PSM数,图C为两种方法甲基化肽段的交集
进一步对两种方法富集到的甲基化肽段及蛋白进行分析后发现,不同方法富集到的甲基化蛋白在GO分类中的功能也有不同,除了被熟知的RNA结合功能的甲基化蛋白是在两种方法都可以被富集到外,SCX富集到的甲基化蛋白更多参与酶活性,而IAP富集到的甲基化蛋白则更多参与基因表达调控相关过程(图D)。除此之外,IAP方法富集的甲基化肽段主要是只含有1个甲基化位点,而SCX富集到的含2、3个甲基化位点的甲基化肽段的比例较IAP更高(图E)。
2、 甲基化肽段的定量结果
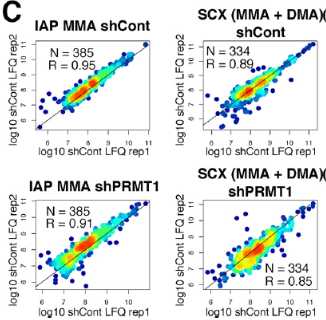
通过maxquant对数据进行label free的方法定量后,首先对定量结果的可重现性进行了分析,结果显示两次生物重复之间的甲基化肽段定量具有较高的相关性(皮尔森相关系数可达到0.85~0.95)(图展示部分结果)。
确定了在两个生物重复之间定量重复性之后,作者探究了在PRMT1蛋白敲除的情况下不同甲基化修饰在表达量水平发生的变化:
1) 精氨酸单甲基化(MMA)肽段的定量分析
PRMT1被报道参与了超过90%的精氨酸ADMA修饰的发生,然而在这篇文章中,作者发现同样对MMA也有影响,通过定量分析,在PRMT1敲除的样本中有61条MMA肽段显著上调和58条MMA肽段显著下调,这119条显著变化的MMA肽段中,只有5条是来自于SCX方法富集,剩余均来自于IAP方法。
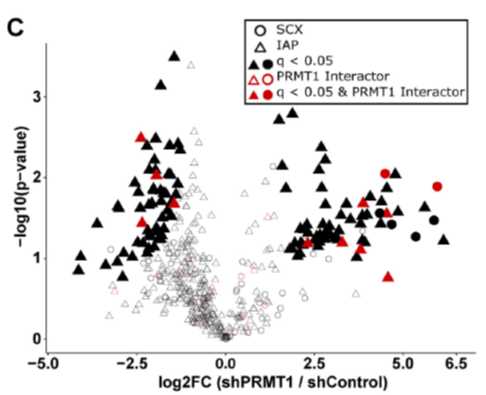
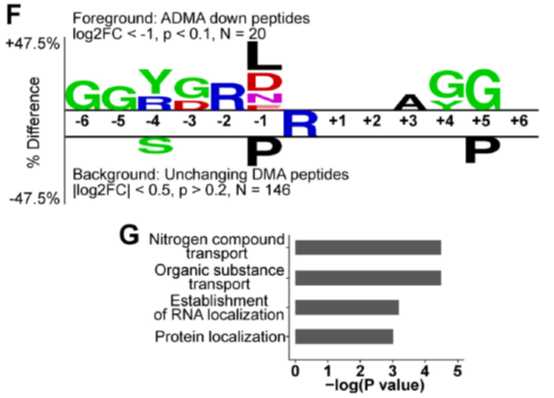
通过对显著变化的MMA肽段的序列分析发现,在发生修饰的精氨酸周围有相对稳定的氨基酸排列模式RGG,且在-4,-2位置更偏向于是丝氨酸,+3位置更偏向于酪氨酸这种侧链具有反应活性的氨基酸。
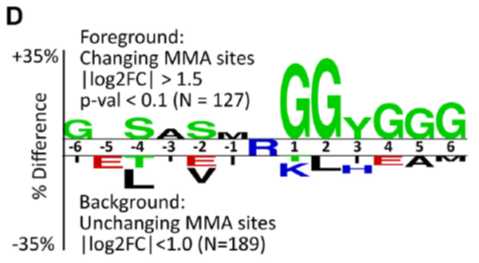
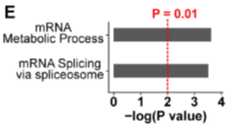
GO分析表明,显著变化的MMA肽段对应的蛋白主要参与mRNA代谢途径和mRNA的剪切。
2)精氨酸二甲基化(DMA)肽段的定量分析
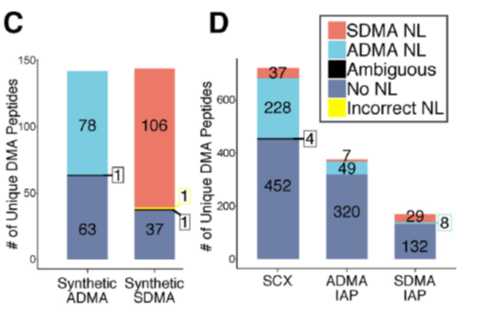
作者首先验证了利用中性丢失的方法鉴定ADMA和SDMA肽段的准确性,他们使用人工合成的ADMA和SDMA数据,使用中性丢失的搜库方法分析后发现,虽然只能鉴定到数据中54.9%的ADMA和73.1%SDMA,但正确率几乎可以达到100%,只鉴定到一条数据中不存在的肽段(图C)。随后他们用这种方法对自己产出的数据进行了分析,结果如图D。
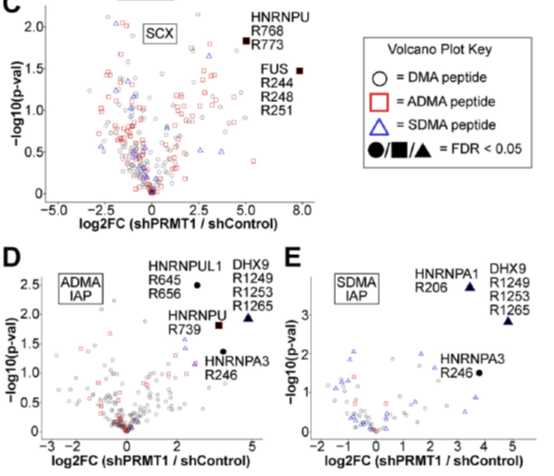
随后对不同富集方法得到的ADMA和SDMA肽段在PRMT1敲除样本中的表达量变化进行了分析,下方火山图依次列出SCX、ADMA\IAP和SDMA\IAP三种富集结果中二甲基化的表达量变化。由图可以看出,虽然有很多肽段在PRMT1敲除样本中的表达量发生剧烈的变化,但通过permutation t test后FDR<0.05的肽段并不多,作者在文章中对此现象做解释。于MMA分析类似,对于DMA的结果,作者同样对发生变化的肽段序列进行了分析和GO功能聚类,不过并没有得到MMA中较为明确的序列特征,但可以看出发生二甲基化表达量变化的蛋白所参与的生物过程于单甲基化是明显不同的。
3、 整合所有精氨酸甲基化定量结果,从而找到PRMT1新的靶标蛋白
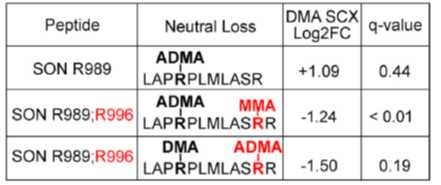
因为已有的研究表面PRMT1是催化二甲基化的产生,作者在这里提出两个条件作为新PRMT1靶标蛋白应该具有的特征:1)因为PRMT1可以同时影响MMA的表达,且在PRMT1敲除后有大量MMA肽段发生上调,所以作者认为只有在PRMT1敲除后ADMA水平下调,但MMA水平上调的蛋白才是只受PRMT1直接作用的靶标;2)因为精氨酸的二甲基化会提高漏切的产生,所以作者重点考虑在产生ADMA修饰的肽段发生漏切和没有漏切的肽段比例;
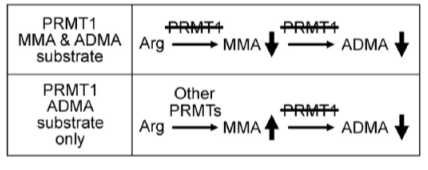
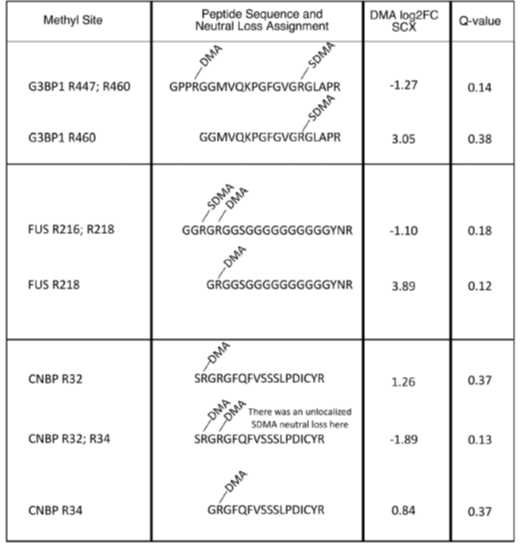
结合以上标准,作者最终发现SON R996具有很高的可信度成为PRMT1的靶标。
四、 文章亮点
个人认为,本篇文章的主要贡献有三点:1)通过比较系统的比较SCX和IAP富集的方法,证明了两种方法在甲基化肽段的富集上具有很好的正交性,提示未来对于甲基化肽段的鉴定应该要考虑结合两种方法同时进行;2)通过中性丢失的搜库方法可以有效的鉴定和区分精氨酸上的对称二甲基化和非对称二甲基化;3)通过对PRMT1蛋白的敲除与未敲除的蛋白质甲基化定量比较,拓宽了对PRMT1新的理解,并找到了可能的新靶标蛋白,为之后要从事类似研究的人员提供了思路。
分享人:张宇星
标签:ola 变化 样本 过程 信号 can max test 使用
原文地址:https://www.cnblogs.com/ilifeiscience/p/12015753.html