标签:dash bottom 适用于 cell 概述 图片 speed default str
期刊名:Mol Cell Proteomics
发表时间:(2019年12月)
IF:4.828
单位:德国Robert Koch 研究所
物种:多种
技术:新蛋白提取和酶解方法
一、 概述:
本文设计了一种仅需要三步——酸化,中和及酶解——就能完成蛋白提取和酶解的实验方法,命名为简单的蛋白提取和酶解法(SPEED)。SPEED使用三氟乙酸(TFA)做裂解液,适用于不同类型的样本,包括难以裂解的样本类型。文章评估了SPEED方法对大肠杆菌,小鼠肝脏和金黄色葡萄球菌的效果,又将该方法与5种不同原理的蛋白提取方法做比较。结果表明SPEED操作简单,成本低廉,蛋白定量重复性高,增加了蛋白质组学的检测深度,在必要时可以大幅度缩短实验时间。SPEED比使用去污剂、离液剂的蛋白提取方法有优势。
二、 研究背景:
基于质谱的蛋白质组学研究多使用bottom-up方法,将蛋白质从样本中提取出来,酶切成肽段,再用质谱仪进行检测。常用的蛋白质提取和酶解方法各有优势和不足之处。例如液态酶解法操作步骤少,方法简便,重复性好,但是不适用于动植物组织、革兰氏阳性菌等较难裂解的样本。本文设计了一种新的样本制备方法SPEED,克服了液态酶解法的缺陷又保留了它的优势。
三、实验设计:

四、研究成果:
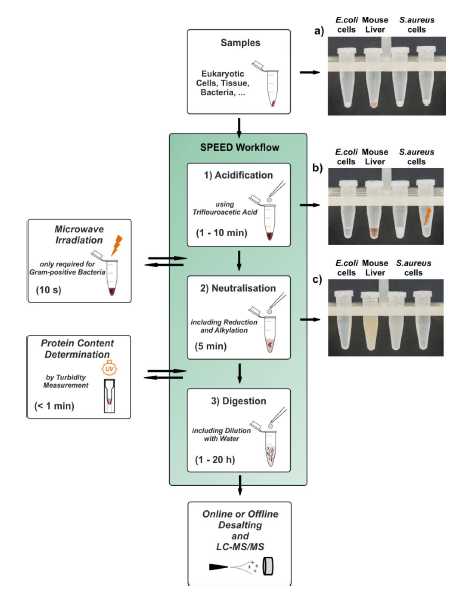
1、SPEED方法流程包括三步,1)用强酸TFA裂解样品,释放出蛋白质,2)用2M的Tris Base中和酸性,3)用胰酶将蛋白消化成肽段。肽段经过线上或线下除盐后,上液相-质谱平台进行检测。TFA能在1min内彻底溶解大肠杆菌细胞, 10min内彻底溶解小鼠肝组织,但金黄葡萄球菌细胞则需在加入TFA后,额外用微波照射10s来溶解。
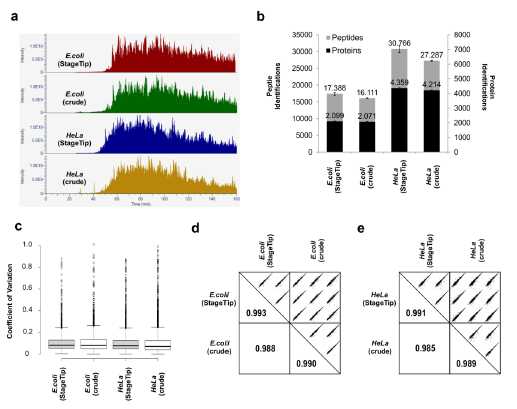
2. 用SPEED方法制备的大肠杆菌和HeLa肽段样品,未除盐样品(在上机时线上除盐)和用stage tip除盐的样品上机检测的结果比较:a)总离子色谱图的强度和出峰时间相近;b)未除盐样品的蛋白鉴定数少1-3%,肽段鉴定数少7-11%;c)-e)蛋白定量重复性,变异系数和Pearson相关系数的结果相近。
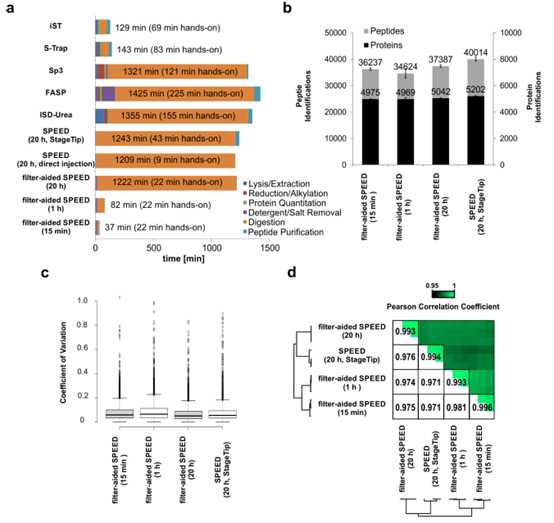
3. 作者在SPEED方法的基础上建立了filter-aided SPEED方法,又将酶解时间从20h减少到了1h或15min,使整个filter-aided SPEED流程的时间减少到82min或37min。作者比较原版SPEED方法(stage tip,酶解20h),filter-aided-酶解20h, 1h, 或15min四种方法,发现使用原版方法得到的蛋白和肽段略多一些。四种方法的蛋白定量稳定性和重复性表现相似。
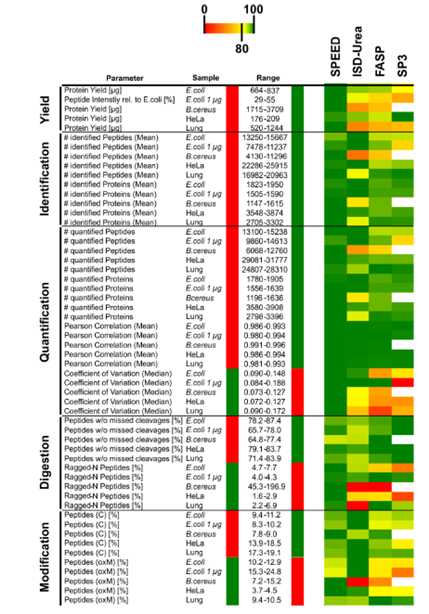
4. 比较SPEED方法与另外三种蛋白提取酶解法在不同类型样本中的效果。文章从蛋白提取率,肽段和蛋白的鉴定数,蛋白定量,酶解,肽段修饰这几个方面对四种方法进行了比较。SPEED的综合表现优秀,主要的优势在于提升了样本的蛋白定量重复性和蛋白质组测序深度。
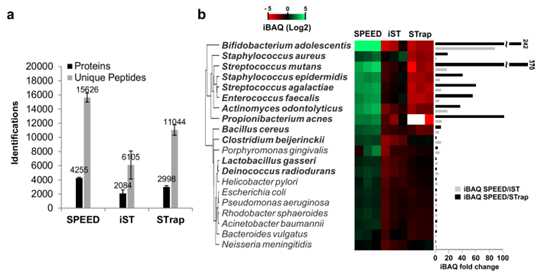
5. 用SPEED方法从包含20株人体微生物的混合样本中提取和酶解蛋白,和iST, STrap两种样本处理方法的效果作比较。SPEED处理的样本,蛋白和肽段鉴定数(a)以及蛋白的绝对定量值(b)都远超另外两种方法,在革兰氏阳性菌中表现明显。这表明用SPEED方法处理难裂解的细菌样本,效果比去污剂和物理裂解的组合方法效果好。
五、文章亮点(结论讨论):
本文设计了一种新的蛋白提取和酶解方法SPEED。不同于广泛的使用去污剂或离液剂与物理裂解的组合,作者另辟蹊径,用强酸溶解细胞膜,释放蛋白,达到裂解的效果。虽然SPEED在破裂细菌细胞壁时需要额外的辅助,作者也没有展示此方法对植物组织的应用,但SPEED在细胞,微生物和动物组织样本中的应用效果很好。
阅读人:李思奇
标签:dash bottom 适用于 cell 概述 图片 speed default str
原文地址:https://www.cnblogs.com/ilifeiscience/p/12053571.html