标签:vmax append width one 数据可视化 集中 result read 包括
定义问题
波士顿房价数据集收集于1978年,包括14个特征和506条数据(每条特征的中文解释暂时忽略)。
分析数据,发现输入的特征属性的度量单位是不统一的,也许需要对数据度量单位进行调整。
导入数据
首先导入项目中需要的类库。
1 #导入类库 2 import numpy as np 3 from numpy import arange 4 from matplotlib import pyplot 5 from pandas import read_csv 6 from pandas import set_option 7 from pandas.plotting import scatter_matrix 8 from sklearn.preprocessing import StandardScaler 9 from sklearn.model_selection import train_test_split 10 from sklearn.model_selection import KFold 11 from sklearn.model_selection import cross_val_score 12 from sklearn.model_selection import GridSearchCV 13 from sklearn.linear_model import LinearRegression 14 from sklearn.linear_model import Lasso 15 from sklearn.linear_model import ElasticNet 16 17 from sklearn.tree import DecisionTreeRegressor 18 from sklearn.neighbors import KNeighborsRegressor 19 from sklearn.svm import SVR 20 from sklearn.pipeline import Pipeline 21 from sklearn.ensemble import RandomForestRegressor 22 from sklearn.ensemble import GradientBoostingRegressor 23 from sklearn.ensemble import ExtraTreesRegressor 24 from sklearn.ensemble import AdaBoostRegressor 25 from sklearn.metrics import mean_squared_error
接下来导入数据集到Python中,在导入数据集时还设定了数据集属性特征的名字。
1 #导入数据 2 filename=‘/home/aistudio/work/housing.csv‘ 3 names=[‘CRIM‘,‘ZN‘,‘INDUS‘,‘CHAS‘,‘NOX‘,‘RM‘,‘AGE‘,‘DIS‘,‘RAD‘,‘TAX‘,‘PRTATIO‘,‘B‘,‘LSTAT‘,‘MEDV‘] 4 data=read_csv(filename,names=names,delim_whitespace=True)
在这里对每一个特征属性设定了一个名称,便于后面的程序中使用它们。因为CSV文件是使用空格键做分隔符,因此读入CSV文件时指定分割符为空格键(delim_whitespace=True)。
理解数据
首先看下数据维度,如数据集中有多少条记录,有多少个数据特征。
1 #数据维度 2 print(data.shape)
(506, 14)
得到506条记录和14个特征属性。
再查看各个特征属性的字段类型:
1 print(data.dtypes)
CRIM float64 ZN float64 INDUS float64 CHAS int64 NOX float64 RM float64 AGE float64 DIS float64 RAD int64 TAX float64 PRTATIO float64 B float64 LSTAT float64 MEDV float64 dtype: object
接下来对数据进行一次简单查看,如前30条记录:
1 set_option(‘display.width‘,200) 2 print(data.head(30))
CRIM ZN INDUS CHAS NOX ... TAX PRTATIO B LSTAT MEDV 0 0.006 18.0 2.31 0 0.538 ... 296.0 15.3 396.90 4.98 24.0 1 0.027 0.0 7.07 0 0.469 ... 242.0 17.8 396.90 9.14 21.6 2 0.027 0.0 7.07 0 0.469 ... 242.0 17.8 392.83 4.03 34.7 3 0.032 0.0 2.18 0 0.458 ... 222.0 18.7 394.63 2.94 33.4 4 0.069 0.0 2.18 0 0.458 ... 222.0 18.7 396.90 5.33 36.2 5 0.030 0.0 2.18 0 0.458 ... 222.0 18.7 394.12 5.21 28.7 6 0.088 12.5 7.87 0 0.524 ... 311.0 15.2 395.60 12.43 22.9 7 0.145 12.5 7.87 0 0.524 ... 311.0 15.2 396.90 19.15 27.1 8 0.211 12.5 7.87 0 0.524 ... 311.0 15.2 386.63 29.93 16.5 9 0.170 12.5 7.87 0 0.524 ... 311.0 15.2 386.71 17.10 18.9 10 0.225 12.5 7.87 0 0.524 ... 311.0 15.2 392.52 20.45 15.0 11 0.117 12.5 7.87 0 0.524 ... 311.0 15.2 396.90 13.27 18.9 12 0.094 12.5 7.87 0 0.524 ... 311.0 15.2 390.50 15.71 21.7 13 0.630 0.0 8.14 0 0.538 ... 307.0 21.0 396.90 8.26 20.4 14 0.638 0.0 8.14 0 0.538 ... 307.0 21.0 380.02 10.26 18.2 15 0.627 0.0 8.14 0 0.538 ... 307.0 21.0 395.62 8.47 19.9 16 1.054 0.0 8.14 0 0.538 ... 307.0 21.0 386.85 6.58 23.1 17 0.784 0.0 8.14 0 0.538 ... 307.0 21.0 386.75 14.67 17.5 18 0.803 0.0 8.14 0 0.538 ... 307.0 21.0 288.99 11.69 20.2 19 0.726 0.0 8.14 0 0.538 ... 307.0 21.0 390.95 11.28 18.2 20 1.252 0.0 8.14 0 0.538 ... 307.0 21.0 376.57 21.02 13.6 21 0.852 0.0 8.14 0 0.538 ... 307.0 21.0 392.53 13.83 19.6 22 1.232 0.0 8.14 0 0.538 ... 307.0 21.0 396.90 18.72 15.2 23 0.988 0.0 8.14 0 0.538 ... 307.0 21.0 394.54 19.88 14.5 24 0.750 0.0 8.14 0 0.538 ... 307.0 21.0 394.33 16.30 15.6 25 0.841 0.0 8.14 0 0.538 ... 307.0 21.0 303.42 16.51 13.9 26 0.672 0.0 8.14 0 0.538 ... 307.0 21.0 376.88 14.81 16.6 27 0.956 0.0 8.14 0 0.538 ... 307.0 21.0 306.38 17.28 14.8 28 0.773 0.0 8.14 0 0.538 ... 307.0 21.0 387.94 12.80 18.4 29 1.002 0.0 8.14 0 0.538 ... 307.0 21.0 380.23 11.98 21.0
[30 rows x 14 columns]
(测试发现,改变显示宽度200到400,显示界面没有变化,暂时不知什么原因?)
数据的描叙性统计信息,如数量,均值,方差等。
1 set_option(‘precision‘,3) 2 print(data.describe())
CRIM ZN INDUS ... B LSTAT MEDV count 506.000 506.000 506.000 ... 506.000 506.000 506.000 mean 3.614 11.364 11.137 ... 356.674 12.653 22.533 std 8.602 23.322 6.860 ... 91.295 7.141 9.197 min 0.006 0.000 0.460 ... 0.320 1.730 5.000 25% 0.082 0.000 5.190 ... 375.377 6.950 17.025 50% 0.257 0.000 9.690 ... 391.440 11.360 21.200 75% 3.677 12.500 18.100 ... 396.225 16.955 25.000 max 88.976 100.000 27.740 ... 396.900 37.970 50.000 [8 rows x 14 columns]
查看数据特征之间两两关联关系,这里查看数据的皮尔荪相关系数。
1 print(data.corr(method=‘pearson‘))
CRIM ZN INDUS CHAS ... PRTATIO B LSTAT MEDV CRIM 1.000 -0.200 0.407 -0.056 ... 0.290 -0.385 0.456 -0.388 ZN -0.200 1.000 -0.534 -0.043 ... -0.392 0.176 -0.413 0.360 INDUS 0.407 -0.534 1.000 0.063 ... 0.383 -0.357 0.604 -0.484 CHAS -0.056 -0.043 0.063 1.000 ... -0.122 0.049 -0.054 0.175 NOX 0.421 -0.517 0.764 0.091 ... 0.189 -0.380 0.591 -0.427 RM -0.219 0.312 -0.392 0.091 ... -0.356 0.128 -0.614 0.695 AGE 0.353 -0.570 0.645 0.087 ... 0.262 -0.274 0.602 -0.377 DIS -0.380 0.664 -0.708 -0.099 ... -0.232 0.292 -0.497 0.250 RAD 0.626 -0.312 0.595 -0.007 ... 0.465 -0.444 0.489 -0.382 TAX 0.583 -0.315 0.721 -0.036 ... 0.461 -0.442 0.544 -0.469 PRTATIO 0.290 -0.392 0.383 -0.122 ... 1.000 -0.177 0.374 -0.508 B -0.385 0.176 -0.357 0.049 ... -0.177 1.000 -0.366 0.333 LSTAT 0.456 -0.413 0.604 -0.054 ... 0.374 -0.366 1.000 -0.738 MEDV -0.388 0.360 -0.484 0.175 ... -0.508 0.333 -0.738 1.000 [14 rows x 14 columns]
通过上面的结果可以看到,有些特征属性之间具有很强关联关系(>0.7或<-0.7):
NOX与INDUS之间皮尔荪相关系数是0.76;
DIS与INDUS之间的皮尔荪相关系数是-0.71.
数据可视化
单一特征图
查看每一个数据特征的单独分布图,多查看几种不同的图表有助于发现更好的方法。
1 #直方图 2 data.hist(sharex=False,sharey=False,xlabelsize=1,ylabelsize=1) 3 pyplot.show()
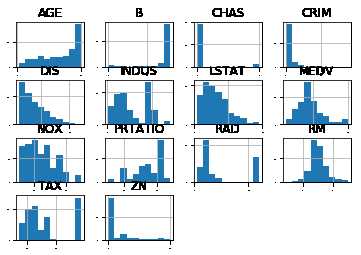
从上图可以看出有些数据呈指数分布,如CRIM,ZN,AGE,B;有些数据呈双峰分布,如RAD和TAX。
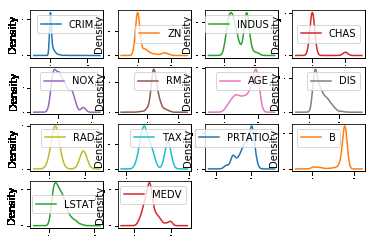
通过密度图可以展示这些数据的特征属性,密度图比直方图更加平滑的展示了这些数据特征。
1 #密度图 2 data.plot(kind=‘density‘,subplots=True,layout=(4,4),sharex=False,fontsize=1) 3 pyplot.show()
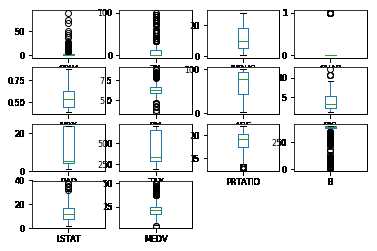
通过箱线图可以查看每一个数据特征的状况,也可以很方便地看出数据分布的偏态程度。
1 #箱线图 2 data.plot(kind=‘box‘,subplots=True,layout=(4,4),sharex=False,sharey=False,fontsize=8) 3 pyplot.show()
多重数据图表
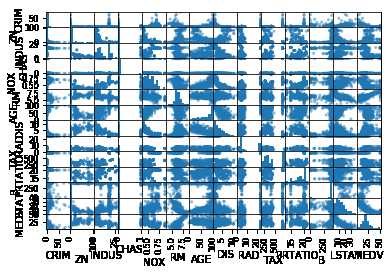
可用多重数据图表来查看不同数据之间的相互影响关系。
1 #散点矩阵图 2 scatter_matrix(data) 3 pyplot.show()
再看下数据相互影响的相关矩阵图。
1 #相关矩阵图 2 fig=pyplot.figure() 3 ax=fig.add_subplot(111) 4 cax=ax.matshow(data.corr(),vmin=-1,vmax=1,interpolation=‘none‘) 5 fig.colorbar(cax) 6 ticks=np.arange(0,14,1) 7 ax.set_xticks(ticks) 8 ax.set_yticks(ticks) 9 ax.set_xticklabels(names) 10 ax.set_yticklabels(names) 11 pyplot.show()
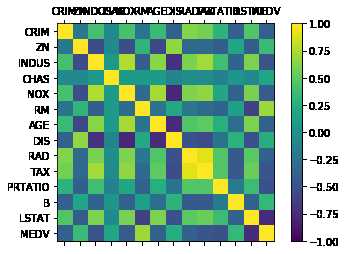
(越接近黄色表示相关性越强)
通过数据的相关性和数据的分布等发现,数据集中的数据结构比较复杂,需要考虑对数据进行转换,以提高模型的准确度。可以尝试:
可以进一步查看数据的可能性分级(离散化),它可以帮助提高决策树的准确度。
分离评估数据集
分离出一个评估数据集需要确保与训练模型的数据集完全隔离,有助于最终判断和报告模型的准确度。这里是2/8开。
1 #分离数据集 2 array=data.values 3 x=array[:,0:13] 4 y=array[:,13] 5 validation_size=0.2 6 seed=7 7 x_train,x_validation,y_train,y_validation=train_test_split(x,y,test_size=validation_size,random_state=seed)
评估算法--原始数据
分析完数据不能立刻选择出哪个算法对需要解决的问题最有效。
直观上认为,由于部分数据的线性分布,线性回归算法和弹性网络回归算法对解决问题可能比较有效。另外,由于数据的离散化,通过决策树算法或支持向量机算法也许可以生成高准确度的模型。
下面采用10折交叉验证来分离数据,通过均方误差来比较算法的准确度。均方误差越趋近于0,算法准确度越高。
1 #评估算法--评估标准 2 num_folds=10 3 seed=7 4 scoring=‘neg_mean_squared_error‘
对原始数据不做任何处理,对算法进行一个评估,形成一个算法的评估基准。这个基准值是对后续算法改善优劣比较的基准值。选择三个线性算法和三个非线性算法来进行比较。
线性算法:线性回归(LR),套索回归(LASSO)和弹性网络回归(EN)
非线性:分类与回归树(CART),支持向量机(SVM)和K近邻(KNN)。
1 models={} 2 models[‘LR‘]=LinearRegression() 3 models[‘LASSO‘]=Lasso() 4 models["EN"]=ElasticNet() 5 models[‘KNN‘]=KNeighborsRegressor() 6 models[‘CART‘]=DecisionTreeRegressor() 7 models[‘SVM‘]=SVR()
对所有的算法使用默认参数,并比较算法的准确度,此处比较的是均方误差的均值和标准方差。
1 result=[] 2 for key in models: 3 kfold=KFold(n_splits=num_folds,random_state=seed) 4 cv_result=cross_val_score(models[key],x_train,y_train,cv=kfold,scoring=scoring) 5 result.append(cv_result) 6 print(‘%s: %f (%f)‘ % (key,cv_result.mean(),cv_result.std()))
从执行的结果来看,线性回归(LR)具有最优的MSE,接下来是分类与回归树算法(CART)。
LR: -21.379856 (9.414264) LASSO: -26.423561 (11.651110) EN: -27.502259 (12.305022) KNN: -41.896488 (13.901688) CART: -25.118465 (13.596293) SVM: -85.518342 (31.994798)
再查看所有的10折交叉分离验证的结果。
1 #评估算法----箱线图 2 fig=pyplot.figure() 3 fig.suptitle(‘Algorithm Comparison‘) 4 ax=fig.add_subplot(111) 5 pyplot.boxplot(result) 6 ax.set_xticklabels(models.keys()) 7 pyplot.show()
执行结果如下图,从图中可以看到,线性算法的分布比较类似,并且K近邻算法的结果分布非常紧凑。
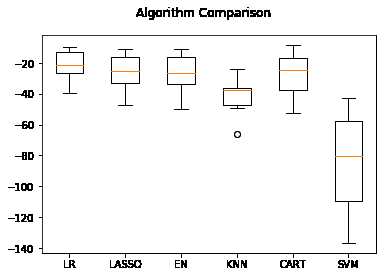
不同的数据度量单位,也许是K近邻算法和支持向量机算法表现不佳的主要原因。
下面将对数据进行正太化处理,再次比较算法的结果。
评估算法----正太化数据
对训练数据集进行数据转换处理,将所有的数据特征值转化成0为中位值,标准差为1的数据。
对数据正太化时,为了防止数据泄漏,采用Pipeline来正太化数据。
为了与前面的结果进行比较,此处采用相同的评估框架来评估算法模型。
1 #评估算法----正太化数据 2 pipelines={} 3 pipelines[‘ScalerLR‘]=Pipeline([(‘Scaler‘,StandardScaler()),(‘LR‘,LinearRegression())]) 4 pipelines[‘ScalerLASSO‘]=Pipeline([(‘Scaler‘,StandardScaler()),(‘LASSO‘,Lasso())]) 5 pipelines[‘ScalerEN‘]=Pipeline([(‘Scaler‘,StandardScaler()),(‘EN‘,ElasticNet())]) 6 pipelines[‘ScalerKNN‘]=Pipeline([(‘Scaler‘,StandardScaler()),(‘KNN‘,KNeighborsRegressor())]) 7 pipelines[‘ScalerCART‘]=Pipeline([(‘Scaler‘,StandardScaler()),(‘CART‘,DecisionTreeRegressor())]) 8 pipelines[‘ScalerSVM‘]=Pipeline([(‘Scaler‘,StandardScaler()),(‘SVM‘,SVR())]) 9 10 results=[] 11 for key in pipelines: 12 kfold=KFold(n_splits=num_folds,random_state=seed) 13 cv_result=cross_val_score(pipelines[key],x_train,y_train,cv=kfold,scoring=scoring) 14 results.append(cv_result) 15 print(‘%s: %f (%f)‘ % (key,cv_result.mean(),cv_result.std()))
执行后,发现K近邻算法具有最优的MES。
ScalerLR: -21.379856 (9.414264) ScalerLASSO: -26.607314 (8.978761) ScalerEN: -27.932372 (10.587490) ScalerKNN: -20.107620 (12.376949) ScalerCART: -27.487674 (12.436629) ScalerSVM: -29.633086 (17.009186)
接下来看一下所有10折交叉分离验证的结果:
1 #评估算法----箱线图 2 fig=pyplot.figure() 3 fig.suptitle(‘Algorithm Comparison‘) 4 ax=fig.add_subplot(111) 5 pyplot.boxplot(results) 6 ax.set_xticklabels(models.keys()) 7 pyplot.show()
从生成箱线图中可以看到K近邻算法具有最优的MSE和最紧凑的数据分布。
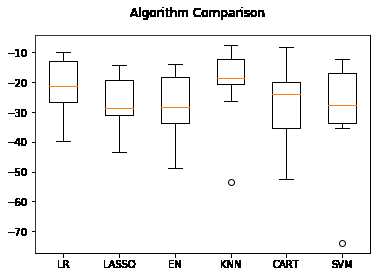
调参改善算法
K近邻算法的默认参数近邻个数(n_neighbors)是5,下面通过网格搜索算法来优化参数。
1 #调参改善算法----KNN 2 scaler=StandardScaler().fit(x_train) 3 rescaledX=scaler.transform(x_train) 4 param_grid={‘n_neighbors‘:[1,3,5,7,9,11,13,15,17,19,21]} 5 model=KNeighborsRegressor() 6 kfold=KFold(n_splits=num_folds,random_state=seed) 7 grid=GridSearchCV(estimator=model,param_grid=param_grid,scoring=scoring,cv=kfold) 8 grid_result=grid.fit(X=rescaledX,y=y_train) 9 10 print(‘最优:%s 使用 %s‘ % (grid_result.best_score_, grid_result.best_params_)) 11 cv_results=zip(grid_result.cv_results_[‘mean_test_score‘],grid_result.cv_results_[‘std_test_score‘],grid_result.cv_results_[‘params‘]) 12 for mean,std,param in cv_results: 13 print(‘%f (%f) with %r‘ % (mean,std,param))
最优结果----K近邻算法的默认参数近邻个数(n_neighbors)是3.
最优:-18.172136963696367 使用 {‘n_neighbors‘: 3}
-20.208663 (15.029652) with {‘n_neighbors‘: 1}
-18.172137 (12.950570) with {‘n_neighbors‘: 3}
-20.131163 (12.203697) with {‘n_neighbors‘: 5}
-20.575845 (12.345886) with {‘n_neighbors‘: 7}
-20.368264 (11.621738) with {‘n_neighbors‘: 9}
-21.009204 (11.610012) with {‘n_neighbors‘: 11}
-21.151809 (11.943318) with {‘n_neighbors‘: 13}
-21.557400 (11.536339) with {‘n_neighbors‘: 15}
-22.789938 (11.566861) with {‘n_neighbors‘: 17}
-23.871873 (11.340389) with {‘n_neighbors‘: 19}
-24.361362 (11.914786) with {‘n_neighbors‘: 21}
集成算法
除了调参之外,提高模型准确度的方法是使用集成算法。
下面会对表现比较好的线性回归、K近邻、分类与回归树进行集成,来看看算法能否提高。
装袋算法:随机森林(RF)和极端随机树(ET)
提升算法:AdaBoost(AB)和随机梯度上升(GBM)
依然采用和前面同样的评估框架和正太化之后的数据来分析相关的算法。
1 #集成算法 2 ensembles={} 3 ensembles[‘ScaledAB‘]=Pipeline([(‘Scaler‘,StandardScaler()),(‘AB‘,AdaBoostRegressor())]) 4 ensembles[‘ScaledAB-KNN‘]=Pipeline([(‘Scaler‘,StandardScaler()),(‘ABKNN‘,AdaBoostRegressor(base_estimator=KNeighborsRegressor(n_neighbors=3)))]) 5 ensembles[‘ScaledAB-LR‘]=Pipeline([(‘Scaler‘,StandardScaler()),(‘ABLR‘,AdaBoostRegressor(LinearRegression()))]) 6 ensembles[‘ScaledRFR‘]=Pipeline([(‘Scaler‘,StandardScaler()),(‘RFR‘,RandomForestRegressor())]) 7 ensembles[‘ScaledETR‘]=Pipeline([(‘Scaler‘,StandardScaler()),(‘ETR‘,ExtraTreesRegressor())]) 8 ensembles[‘ScaledGBR‘]=Pipeline([(‘Scaler‘,StandardScaler()),(‘RBR‘,GradientBoostingRegressor())]) 9 10 results=[] 11 for key in ensembles: 12 kfold=KFold(n_splits=num_folds,random_state=seed) 13 cv_result=cross_val_score(ensembles[key],x_train,y_train,cv=kfold,scoring=scoring) 14 results.append(cv_result) 15 print(‘%s: %f (%f)‘ % (key,cv_result.mean(),cv_result.std()))
与前面的线性算法和非线性算法相比,这次准确度都有了较大的提高。
ScaledAB: -15.396665 (6.887380) ScaledAB-KNN: -15.897982 (9.477002) ScaledAB-LR: -24.463893 (10.070492)
ScaledRFR: -13.172716 (5.511913) ScaledETR: -10.210106 (5.516494)
ScaledGBR: -10.099802 (4.388978)
接下来通过箱线图看一下算法在10折交叉验证中均方误差的分布情况。
1 #集成算法----箱线图 2 fig=pyplot.figure() 3 fig.suptitle(‘Algorithm Comparison‘) 4 ax=fig.add_subplot(111) 5 pyplot.boxplot(results) 6 ax.set_xticklabels(ensembles.keys()) 7 pyplot.show()
执行结果如下图,随机梯度上升算法和极端随机树算法具有较高的中位值和分布状况。
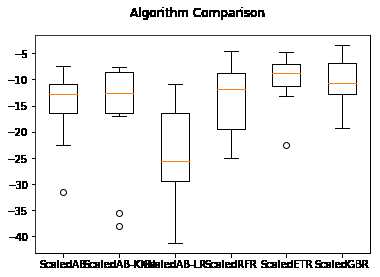
集成算法调参
集成算法都有一个参数n_estimators,这是一个很好的可以用来调整的参数,会带来更准确的结果。下面对随机梯度上升(GBM)和极端随机树(ET)进行调参。再次比较这两个算法的准确度,来确定最终的算法模型。
1 #集成算法GBM--调参 2 caler=StandardScaler().fit(x_train) 3 rescaledX=scaler.transform(x_train) 4 param_grid={‘n_estimators‘:[10,50,100,200,300,400,500,600,700,800,900]} 5 model=GradientBoostingRegressor() 6 kfold=KFold(n_splits=num_folds,random_state=seed) 7 grid=GridSearchCV(estimator=model,param_grid=param_grid,scoring=scoring,cv=kfold) 8 grid_result=grid.fit(X=rescaledX,y=y_train) 9 print(‘最优:%s 使用%s‘ % (grid_result.best_score_,grid_result.best_params_)) 10 11 #集成算法ET--调参 12 caler=StandardScaler().fit(x_train) 13 rescaledX=scaler.transform(x_train) 14 param_grid={‘n_estimators‘:[5,10,20,30,40,50,60,70,80,90,100]} 15 model=ExtraTreesRegressor() 16 kfold=KFold(n_splits=num_folds,random_state=seed) 17 grid=GridSearchCV(estimator=model,param_grid=param_grid,scoring=scoring,cv=kfold) 18 grid_result=grid.fit(X=rescaledX,y=y_train) 19 print(‘最优:%s 使用%s‘ % (grid_result.best_score_,grid_result.best_params_))
对于随机梯度上升(GBM)算法来说,最优的n_estimators=400,而对于极端随机树(ET)来说,最优的n_estimators=90,且,ET稍优于GBM,因此采用极端随机树算法来训练最终模型。
最优:-9.26198126367106 使用{‘n_estimators‘: 400}
最优:-8.999967152548582 使用{‘n_estimators‘: 90}
小技巧,当最优参数是param_grid的边界值时,有必要调整param_grid进行下一次调参。
1 #训练模型 2 caler=StandardScaler().fit(x_train) 3 rescaledX=scaler.transform(x_train) 4 gbr=ExtraTreesRegressor(n_estimators=90) 5 gbr.fit(X=rescaledX,y=y_train)
再通过评估数据集来评估算法的准确度。
1 #评估算法模型 2 rescaledX_validation=scaler.transform(x_validation) 3 predictions=gbr.predict(rescaledX_validation) 4 print(mean_squared_error(y_validation,predictions))
得到执行结果:
13.974640426046953
标签:vmax append width one 数据可视化 集中 result read 包括
原文地址:https://www.cnblogs.com/yuzaihuan/p/12963355.html