标签:今天 类型 方法 感受 深度 中心 输入 bsp 描述
原文链接
扫码关注下方公众号:"Python编程与深度学习",领取配套学习资源,并有不定时深度学习相关文章及代码分享。

今天分享一篇发表在MICCAI 2019上的关于医学影像处理中小器官分割的论文:FocusNet: Imbalanced Large and Small Organ Segmentation with an End-to-End Deep Neural Network for Head and Neck CT Images (原文链接:[1])。
1 研究背景
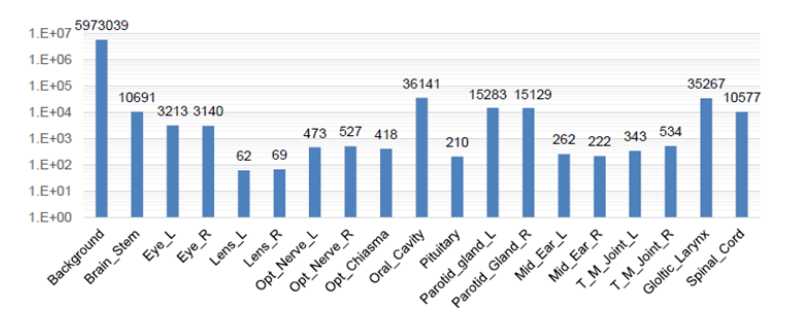
器官分割是医学影像处理中的一类主要任务,现在主流的方法是采用卷积神经网络进行处理。但是不同的器官之间的大小具有很大的差距,如上图所示,晶状体(最小的器官)只占整个3D空间的0.0028%,而腮腺所占区域大约是晶状体的250倍,不同器官之间所占空间的比例是不平衡的。然而,现有的分割神经网络并没有针对这个问题进行优化,这些网络一般能对大器官产生准确的分割图,但对小器官的分割精度往往是不高的。
为了解决上述大小器官分割任务中存在的占比不平衡问题,这篇文章模拟了专家标注的过程。作者发现,对于大器官,专家通常在正常视图下进行标注,而对于小器官,他们先在正常视图下找到对应的位置,然后放大进行标记。受此启发,这篇文章提出了一种新颖的端到端三维卷积神经网络 (FocusNet),同时对大器官和小器官产生精确的分割。
2 方法
2.1 整体流程
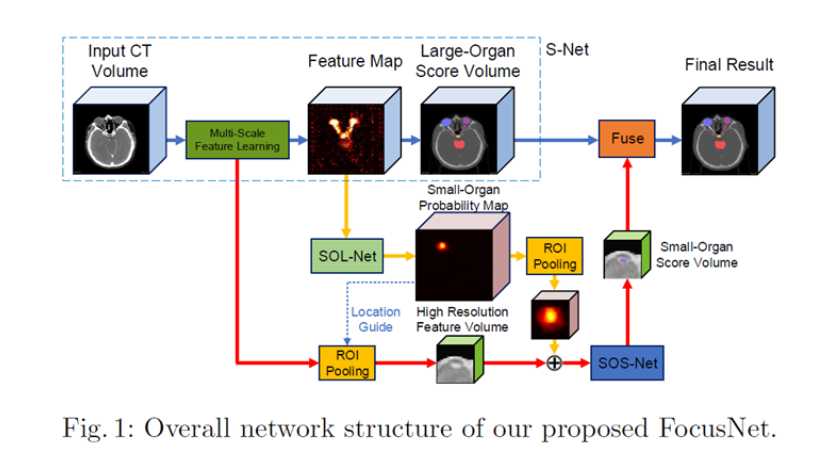
如上图(Fig.1)所示,FocusNet接受3D的CT扫描图作为输入,其具体流程为:
- 通过主分割网络 (S-Net)对大器官预测分割图
- 同时以S-Net的最后一层特征作为SOL-Net的输入,预测小器官的大概位置
- 根据第2步预测的位置截取对应区域的多尺度特征和高分辨率特征作为输入(这里采用ROI Pooling确保输入特征维度的一致性),采用SOS-Net预测小器官的分割图
- 融合第1步和第3步的预测结果得到最终的预测
通过上述具体流程的描述,可以将FocusNet分为三个主要子网络:S-Net、SOL-Net和SOS-Net,接下来将对这几个部分分别进行详细的介绍。
2.2 主分割网络 (S-Net)
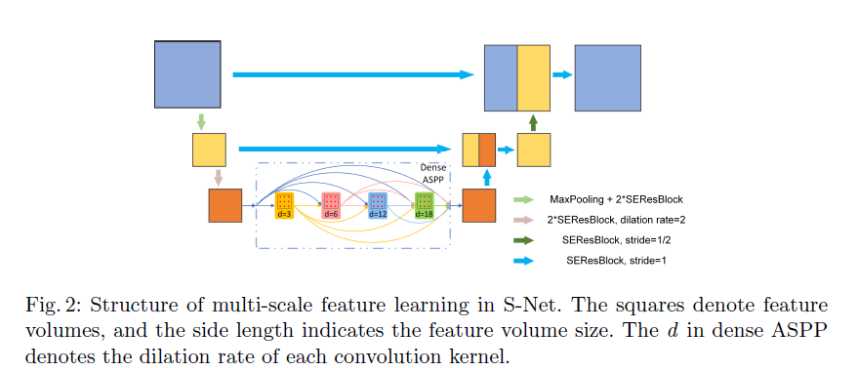
文中通过主分割网络预测大器官的分割图,如上图所示(Fig.2)。作者在文中说明了直接采用3D U-Net在这个任务上的效果并不好,原因有:
- 3D U-Net存在4次下采样过程,然而过多的下采样会导致高分辨率信息的丢失,这将非常不利于对只占有少数体素的小器官进行预测
- 3D U-Net只能通过下采样的方式捕获固定尺度的特征,这限制了它的表示能力(即没有采用类似ASPP模块)
文中提出了S-Net作为主分割网络来避免上述的局限性。S-Net的设置如下:
- 输入:3D的CT扫描图
- 输出:大器官的预测分割图
- 结构: 如Fig.2绿色箭头所示,S-Net只采用一次下采样,同时为了捕获多尺度的特征信息并且扩充感受野,采用DenseASPP模块
- 损失函数:Focal Loss+Dice Loss
2.3 小器官定位网络 (Small-Organ Localization Network, SOL-Net)
通过S-Net已经可以预测大器官的分割图了,接下来需要通过SOL-Net模拟专家标注中定位小器官位置的过程。SOL-Net的设置如下:
- 输入:S-Net的最后一层解码器的特征
- 输出:小器官中心位置的概率热图 (heatmap),每一个小器官都有一个单独的热图(假设小器官类型数量是$K$,那么预测结果的通道数为$K$)
- 结构:由2个Squeeze-and-Excitation Residual Blocks (SEResBlock)和一个1x1x1卷积层组成,最后一层的激活函数为Sigmoid
- 损失函数:MSE Loss
2.4 小器官分割网络 (Small-Organ Segmentation Network, SOS-Net)
通过SOL-Net获得了小器官的位置之后,需要聚焦到相应的小块区域,再用SOS-Net在这个小区域内对小器官进行精确分割。SOS-Net的设置如下:
- 输入:SOL-Net的输出取概率值最大的位置作为各类小器官的位置,第一部分输入为采用ROI-Pooling在SOL-Net的输出中提取各类小器官位置周围的特征,第二部分输入为采用ROI-Pooling提取S-Net解码器 (decoder)最后一层多尺度特征以及S-Net编码器 (encoder)第一层特征各类小器官位置周围的特征,两部分输入相加起来作为输入
- 输出:各类小器官的分割图
- 结构:由2个Squeeze-and-Excitation Residual Blocks (SEResBlock)和一个1x1x1卷积层组成
3 实验结果
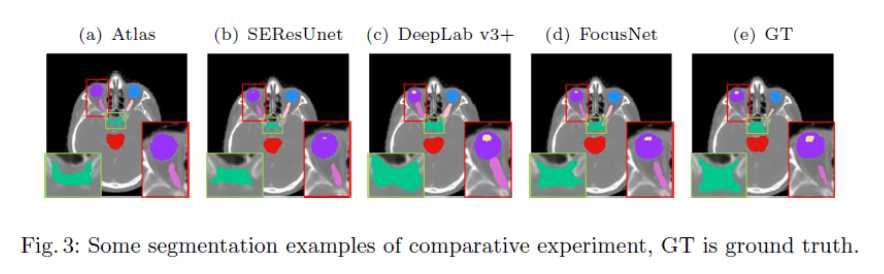
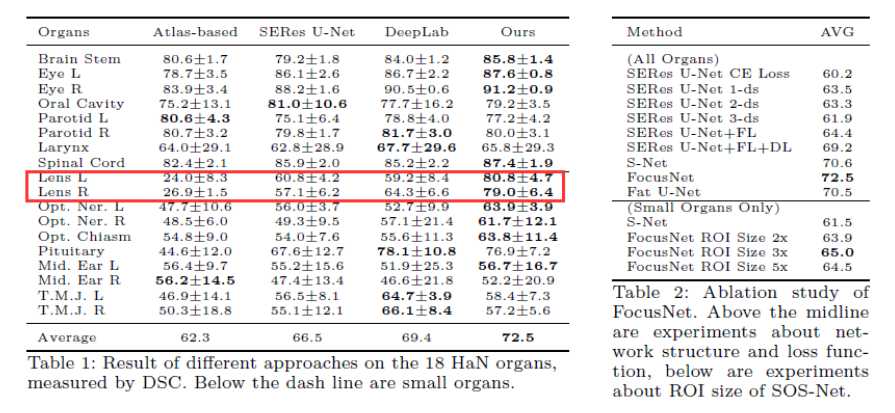
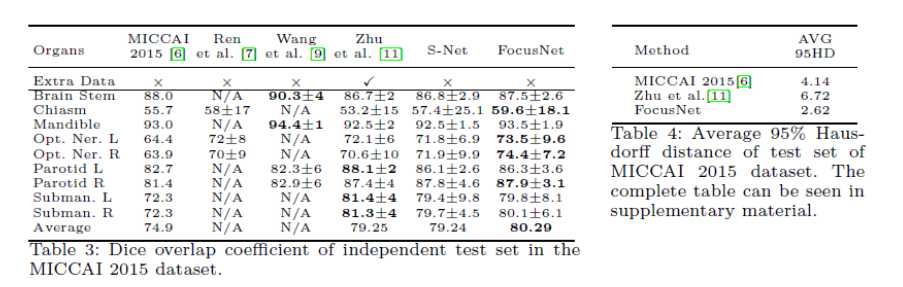
这里我只给出论文中的部分实验结果,具体的实验结果分析以及实验和参数的设置请看原文。
4 参考资料
[1] https://arxiv.org/pdf/1907.12056
FocusNet: Imbalanced Large and Small Organ Segmentation with an End-to-End Deep Neural Network for Head and Neck CT Images(理解)
标签:今天 类型 方法 感受 深度 中心 输入 bsp 描述
原文地址:https://www.cnblogs.com/CZiFan/p/13175914.html






