标签:
ENIGMA是一个国际合作项目,参与人员遍布全球,主要目的是整合全球各方人员手中的脑影像数据来探究脑疾病与大脑的解剖结构和功能性之间的关系。ENIGMA整理了大量有关脑影像分析和处理的软件,并提供了比较规范的处理脑影像数据的一般步骤,其中,ENIGMA DTI Protocol为处理DTI图像的标准步骤。本文在该协议的基础上更为详细地介绍了如何对DTI进行FA值的统计分析。有关ENIGMA DTI Protocol的详细介绍参见:http://enigma.ini.usc.edu/protocols/dti-protocols/
本文的目的是通过对DTI进行处理和分析,统计出每个病人的每个大脑区域的FA值,处理过程主要包括三个模块:
ENIGMA-DTI Skeletonization
ROI extraction from FA images
最终得到的结果为一个.csv文件,内容如下:
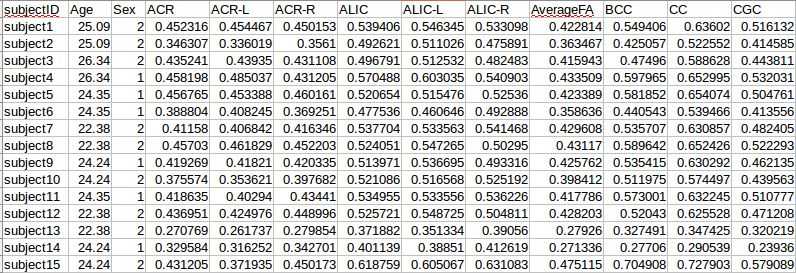
假设当前共有89个病人的数据,每个病人的数据都包含T1,T2,DTI等多种数据,且这些数据都放置在同一文件夹下。
RadiAnt DICOM Viewer可用于查看dcm文件,并按需要导出相应的文件。一个病人通常含有多种脑影像数据,例如T1, T2,DTI等。当这些数据全部置于同一文件夹下时,肉眼难以分辨出数据类型。由于我们的数据中每个病人的所有类型的数据都在同一文件夹下,因此这里利用该软件实现文件的分类。操作步骤:用该软件加载一个病人的数据(选择相应的文件夹即可),如下图所示,左边显示该病人包含几个不同类型的数据(这里为7个),并且显示了每个数据的类型。选择import image,弹出如下所示对话框,选择Current series以及DICOM,软件将导出当前选中的序列,如果选择All opened series,软件将会把所有的序列导出到同一文件夹下。
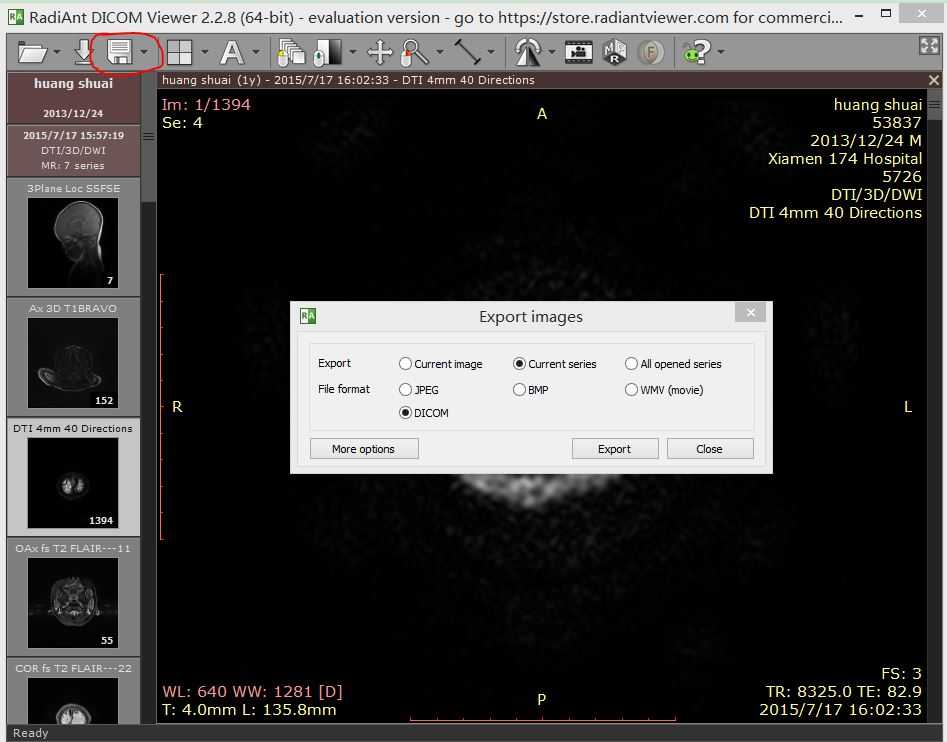
依次选择每个序列,并按上述操作导出每个序列,这样便可实现文件的分类,如下图所示:

PS:如果病人的数据不需要分类整理可跳过这步
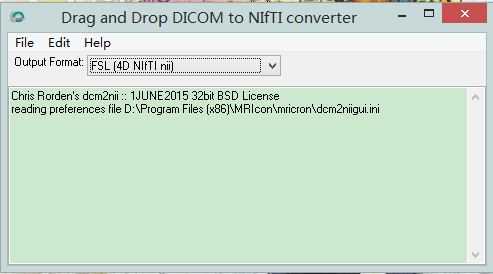
dcm2nii可将dcm文件转化为nii格式的文件,打开dcm2nii,并将需要转换的文件夹直接拖入 dcm2nii即可。
需要处理的总共有89个病人数据,为了方便批处理,将包含病人数据的所有的文件夹名修改为subject*的形式。为了后面采用dtifit计算FA值,修改每个文件夹下的bvec与bval的文件名(.bvec修改为original.bvec,.bval修改为bvals),shell代码如下所示:
for subj in {1..89}
do
mv ./subject${subj}/*.bvec ./subject${subj}/original.bvec
mv ./subject${subj}/*.bval ./subject${subj}/bvals
done
将上述代码保存到rename.sh文件中,在终端调用rename.sh文件:
chmod +x ./rename.sh ./rename.sh
注意这里默认rename.sh与各subject放在同一文件夹内。
在终端输入Fdt,在左上角选择Eddy current correction,然后选择相应的文件即可。
这一步将输出两个文件:
用Fdt进行eddy_correct之后需要调整bvecs文件,新建两个文件分别命名为rotate_bvecs_core.sh与rotateBvecs.sh,代码如下所示:
rotateBvecs.sh
#!/bin/bash for subj in {1..89} do sh rotate_bvecs_core.sh ./subject${subj}/original.bvec ./subject${subj}/bvecs ./subject${subj}/data.ecclog done
#!/bin/sh #Originally written by Saad Jbabdi # University of Oxford, FMRIB Centre # and posted on JISCMail FSL Archives Feb 3 2012 # modified by Neda Jahanshad (USC/ENIGMA-DTI) # to take in vertically oriented bvec files if [ "$3" == "" ] ; then echo "Usage: <original bvecs> <rotated bvecs> <ecclog>" echo "" echo "<ecclog> is the output log file from ecc" echo "" exit 1; fi i=$1 o=$2 ecclog=$3 if [ ! -e $1 ] ; then echo "Source bvecs $1 does not exist!" exit 1 fi if [ ! -e $ecclog ]; then echo "Ecc log file $3 does not exist!" exit 1 fi nline=$(cat $i | wc -l ) if [ $nline -gt 3 ] then echo "the file is vertical and will be transposed" awk ‘ { for (k=1; k<=NF; k++) { a[NR,k] = $k } } NF>p { p = NF } END { for(j=1; j<=p; j++) { str=a[1,j] for(k=2; k<=NR; k++){ str=str" "a[k,j]; } print str } }‘ $i > ${i}_horizontal i=${i}_horizontal fi ii=1 rm -f $o tmpo=${o}$$ cat ${ecclog} | while read line; do echo $ii if [ "$line" == "" ];then break;fi read line; read line; read line; echo $line > $tmpo read line echo $line >> $tmpo read line echo $line >> $tmpo read line echo $line >> $tmpo read line m11=`avscale $tmpo | grep Rotation -A 1 | tail -n 1| awk ‘{print $1}‘` m12=`avscale $tmpo | grep Rotation -A 1 | tail -n 1| awk ‘{print $2}‘` m13=`avscale $tmpo | grep Rotation -A 1 | tail -n 1| awk ‘{print $3}‘` m21=`avscale $tmpo | grep Rotation -A 2 | tail -n 1| awk ‘{print $1}‘` m22=`avscale $tmpo | grep Rotation -A 2 | tail -n 1| awk ‘{print $2}‘` m23=`avscale $tmpo | grep Rotation -A 2 | tail -n 1| awk ‘{print $3}‘` m31=`avscale $tmpo | grep Rotation -A 3 | tail -n 1| awk ‘{print $1}‘` m32=`avscale $tmpo | grep Rotation -A 3 | tail -n 1| awk ‘{print $2}‘` m33=`avscale $tmpo | grep Rotation -A 3 | tail -n 1| awk ‘{print $3}‘` X=`cat $i | awk -v x=$ii ‘{print $x}‘ | head -n 1 | tail -n 1 | awk -F"E" ‘BEGIN{OFMT="%10.10f"} {print $1 * (10 ^ $2)}‘ ` Y=`cat $i | awk -v x=$ii ‘{print $x}‘ | head -n 2 | tail -n 1 | awk -F"E" ‘BEGIN{OFMT="%10.10f"} {print $1 * (10 ^ $2)}‘ ` Z=`cat $i | awk -v x=$ii ‘{print $x}‘ | head -n 3 | tail -n 1 | awk -F"E" ‘BEGIN{OFMT="%10.10f"} {print $1 * (10 ^ $2)}‘ ` rX=`echo "scale=7; ($m11 * $X) + ($m12 * $Y) + ($m13 * $Z)" | bc -l` rY=`echo "scale=7; ($m21 * $X) + ($m22 * $Y) + ($m23 * $Z)" | bc -l` rZ=`echo "scale=7; ($m31 * $X) + ($m32 * $Y) + ($m33 * $Z)" | bc -l` if [ "$ii" -eq 1 ];then echo $rX > $o;echo $rY >> $o;echo $rZ >> $o else cp $o $tmpo (echo $rX;echo $rY;echo $rZ) | paste $tmpo - > $o fi let "ii+=1" done rm -f $tmpo
在终端调用rotateBvecs.sh即可。
在终端输入fsl,然后选择BET brain extraction,在左侧的对话框中需要指定输入图像,这里为data.nii.gz。然后再advanced options中选中前两项,第一项为生成去掉颅骨的图像,第二项生成一个mask(这里需要的是第二项,未免以后需要,这里连同第一项一起生成)。
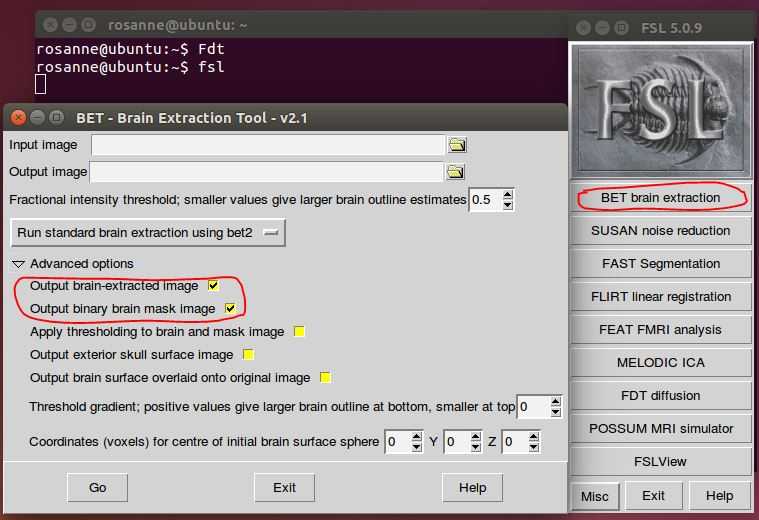
为了实现批量处理,需要在终端中使用命令来调用bet。新建一个文件命名为runBet.sh,代码如下所示:
for subj in {1..89} do ${FSLDIR}/bin/bet ./subject${subj}/data.nii.gz ./subject${subj}/data_brain.nii.gz -m mv ./subject${subj}/data_brain_mask.nii.gz ./subject${subj}/nodif_brain_mask.nii.gz done
(FSLDIR为设置fsl的安装路径,在安装fsl时指定)
bet命令的详细解释参见:http://fsl.fmrib.ox.ac.uk/fsl/fslwiki/BET/UserGuide
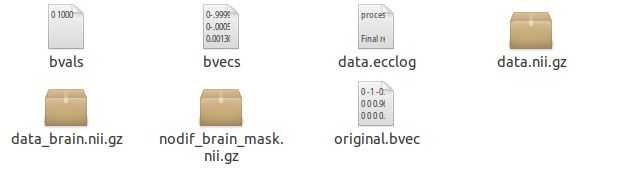
此时,每个subject的文件夹中应该包含以下几个数据:
由于这一步骤需要额外的数据,而我们并没有这样的数据,且我们的数据本身质量较好,因此这里跳过(如果图像本身质量较差则不可跳过)。处理方法参见下图:

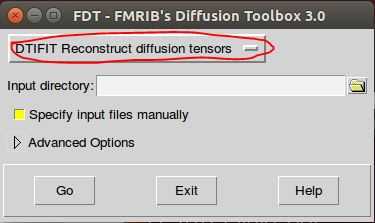
处理单个文件时,可直接用带界面的Fdt处理。在终端输入Fdt,然后在左上角的下拉框中选择dtifit选项,指定待处理数据所在的文件夹,注意文件夹下必须包含data.nii.gz, nodif_brain_mask.nii.gz, bvals, bvecs这四个文件,且文件名必须一致。
若要实现批量处理,需用dtifit命令。新建一个文件命名为runDtifit.sh,代码如下:
for subj in {1..89} do ${FSLDIR}/bin/dtifit -k ./subject${subj}/data.nii.gz -m ./subject${subj}/nodif_brain_mask.nii.gz -r ./subject${subj}/bvecs -b ./subject${subj}/bvals -o ./subject${subj}/dti done
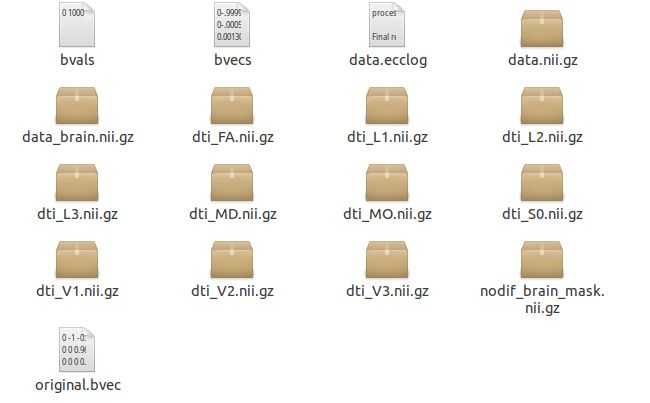
dtifit处理完后,文件夹内应包含一下文件:
标签:
原文地址:http://www.cnblogs.com/happygirl-zjj/p/5096824.html