标签:return turn mem class ons 计算 using pre scan
大家都知道,基因可以看作一个碱基对序列。它包含了4种核苷酸,简记作A,C,G,T。生物学家正致力于寻找人类基因的功能,以利用于诊断疾病和发明药物。
在一个人类基因工作组的任务中,生物学家研究的是:两个基因的相似程度。因为这个研究对疾病的治疗有着非同寻常的作用。
两个基因的相似度的计算方法如下:
对于两个已知基因,例如AGTGATG和GTTAG,将它们的碱基互相对应。当然,中间可以加入一些空碱基-,例如:
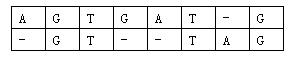
这样,两个基因之间的相似度就可以用碱基之间相似度的总和来描述,碱基之间的相似度如下表所示:
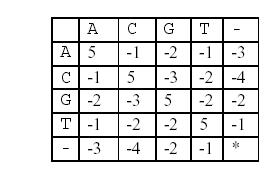
那么相似度就是:(-3)+5+5+(-2)+(-3)+5+(-3)+5=9。因为两个基因的对应方法不唯一,例如又有:
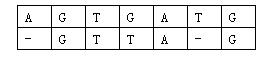
相似度为:(-3)+5+5+(-2)+5+(-1)+5=14。规定两个基因的相似度为所有对应方法中,相似度最大的那个。
输入格式:
共两行。每行首先是一个整数,表示基因的长度;隔一个空格后是一个基因序列,序列中只含A,C,G,T四个字母。1<=序列的长度<=100。
输出格式:
仅一行,即输入基因的相似度。
7 AGTGATG 5 GTTAG
14
#include<stdio.h> #include<string.h> #include<memory.h> using namespace std; inline int dmax(int x,int y){ if(x>y) return x; return y; } inline int pro(char c){ if(c==‘A‘) return 1; if(c==‘C‘) return 2; if(c==‘G‘) return 3; if(c==‘T‘) return 4; return 5; } const int N=101; const int sim[7][7]={ {0,0,0,0,0,0}, {0,5,-1,-2,-1,-3}, {0,-1,5,-3,-2,-4}, {0,-2,-3,5,-2,-2}, {0,-1,-2,-2,5,-1}, {0,-3,-4,-2,-1,0} }; int ls,lt,f[N][N],s[N],t[N]; char c1[N],c2[N]; int main(){ scanf("%d%s%d%s",&ls,c1,<,c2); for(int i=0;i<ls;i++) s[i+1]=pro(c1[i]), f[i+1][0]=sim[5][s[i+1]]+f[i][0]; for(int j=0;j<lt;j++) t[j+1]=pro(c2[j]), f[0][j+1]=sim[5][t[j+1]]+f[0][j]; for(int i=1;i<=ls;i++) for(int j=1;j<=lt;j++) f[i][j]=dmax(f[i-1][j-1]+sim[s[i]][t[j]],dmax(f[i-1][j]+sim[5][s[i]],f[i][j-1]+sim[5][t[j]])); printf("%d\n",f[ls][lt]); return 0; }
标签:return turn mem class ons 计算 using pre scan
原文地址:http://www.cnblogs.com/keshuqi/p/6072379.html