标签:引用 max 通用 1.2 需要 rac enter 不规则 bic
说来惭愧,很久没有更新自己的博客了。期间个人生活经历了很多变故,心理上的打击尤甚。加之没有取得好的科研成果,痛定思痛,还是下苦功夫多多学习。
最近对比验证各种方法的插值精度,用到了R语言地统计学包,由于没有中文文档,故自己干脆将其翻译了出来。欢迎转载引用,但请注明出处http://www.cnblogs.com/jingnianq/p/6606159.html 。
geoR提供了使用软件R进行地统计数据分析的功能。
本文档介绍了一些(但不是全部的)包的功能。 目的是让读者熟悉geoR的数据分析命令,并显示一些可以生成的图形输出。 这里使用的命令仅仅是说明性的,提供了包处理的基本示例。
我们没有试图对这些数据进行明确的分析。
接下来的R命令如下所示
typewriter fonts like this.
通常函数调用使用默认参数,我们鼓励用户检查函数的其他参数。 例如,要查看函数variog类型的所有参数:
args(variog)
本页显示的命令也可以在geoRintro.R文件中找到。
参考geoR文档,了解包geoR中包含的功能的更多详细信息。
启动R会话后,使用以下命令加载geoR:
library(geoR)
如果软件包的安装目录是R软件包的默认位置,请键入:
library(geoR, lib.loc="PATH_TO_geoR")
其中“PATH_TO_geoR”是安装geoR的目录的路径。 如果包装正确加载,将显示以下消息:
------------------------------------------------
geoR: functions for geostatistical data analysis
geoR version 1.3-17 is now loaded
------------------------------------------------
通常,数据被存储为类“geodata”的对象(列表)。此类的对象至少包含数据位置和属性值。
单击以获取有关如何从ASCII(文本)文件读取数据的信息。
有关如何导入/转换数据以及类“geodata”的定义的更多信息,请参考函数as.geodata和read.geodata的文档。 对于本文档中包含的示例,我们使用geoR分布中包含的数据集s100。
要加载此数据类型:
data(s100)
获得数据的快速摘要可以通过输入:
summary(s100)
这将返回坐标和数据值的摘要
$coords.summary
[,1] [,2]
min 0.005638006 0.01091027
max 0.983920544 0.99124979
$data.summary
Min. 1st Qu. Median Mean 3rd Qu. Max.
-1.1680 0.2730 1.1050 0.9307 1.6100 2.8680
如果covariate, borders或者 units.m存在于geodata中,则将会一起汇总。
函数plot.geodata显示一个2 x 2的图表,包括数据位置(上图)和数据与坐标(data versus coordinates,底部图)。 对于类 “geodata”的对象,绘图由该命令完成:
plot(s100)
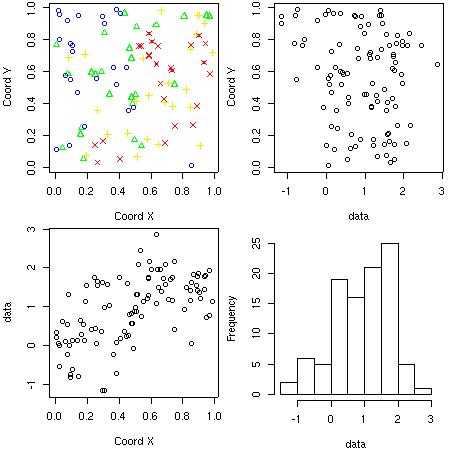
请注意,右上图是使用包scatterplot3d生成的。 如果没有安装此软件包,数据直方图将取代该图。
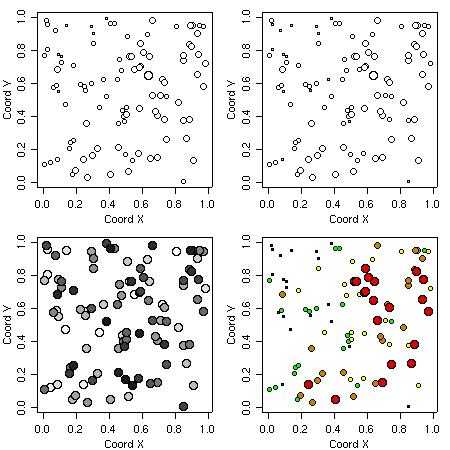
函数point.geodata生成一个显示数据位置的图, 或者将数据位置点添加到当前图。
可以选择指定点大小,图案和颜色,可以将其设置为与数据值或指定的分位数成比例。
图形输出的命令和相应的绘图方法如下所示。
我们开始保存当前的图形参数。
par.ori <- par(no.readonly = TRUE)
par(mfrow = c(2,2))
points(s100, xlab = "Coord X", ylab = "Coord Y")
points(s100, xlab = "Coord X", ylab = "Coord Y", pt.divide = "rank.prop")
points(s100, xlab = "Coord X", ylab = "Coord Y", cex.max = 1.7,
col = gray(seq(1, 0.1, l=100)), pt.divide = "equal")
points(s100, pt.divide = "quintile", xlab = "Coord X", ylab = "Coord Y")
par(par.ori)
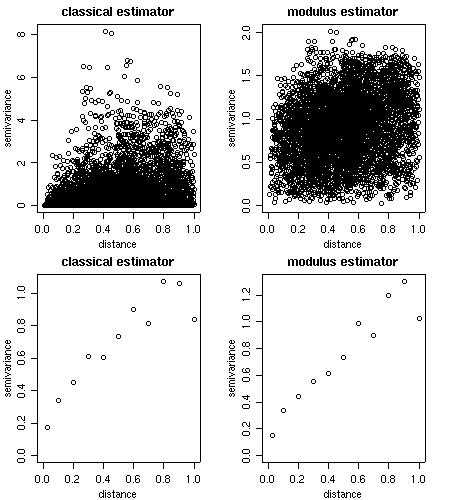
使用函数variog计算经验变差函数。也可以选择经典或modulus估计。
运行结果可以返回变差云、分级或平滑变差函数。
cloud1 <- variog(s100, option = "cloud", max.dist=1)
cloud2 <- variog(s100, option = "cloud", estimator.type = "modulus", max.dist=1)
bin1 <- variog(s100, uvec=seq(0,1,l=11))
bin2 <- variog(s100, uvec=seq(0,1,l=11), estimator.type= "modulus")
par(mfrow=c(2,2))
plot(cloud1, main = "classical estimator")
plot(cloud2, main = "modulus estimator")
plot(bin1, main = "classical estimator")
plot(bin2, main = "modulus estimator")
par(par.ori)
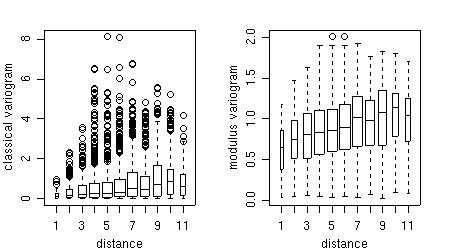
bin1 <- variog(s100,uvec = seq(0,1,l=11), bin.cloud = T)
bin2 <- variog(s100,uvec = seq(0,1,l=11), estimator.type = "modulus", bin.cloud = T)
par(mfrow = c(1,2))
plot(bin1, bin.cloud = T, main = "classical estimator")
plot(bin2, bin.cloud = T, main = "modulus estimator")
par(par.ori)
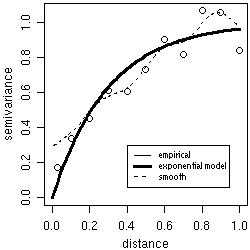
可以绘制并直观比较理论和经验变异函数。 例如,下图显示了用于模拟数据s100的理论变差函数模型和两个估计模型。
bin1 <- variog(s100, uvec = seq(0,1,l=11))
plot(bin1)
lines.variomodel(cov.model = "exp", cov.pars = c(1,0.3), nugget = 0, max.dist = 1, lwd = 3)
smooth <- variog(s100, option = "smooth", max.dist = 1, n.points = 100, kernel = "normal", band = 0.2)
lines(smooth, type ="l", lty = 2)
legend(0.4, 0.3, c("empirical", "exponential model", "smoothed"), lty = c(1,1,2), lwd = c(1,3,1))
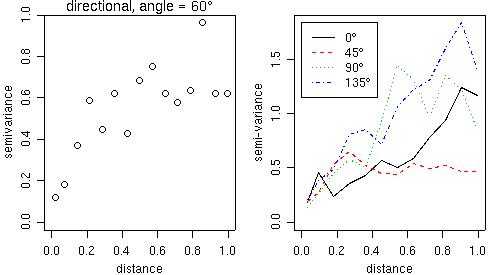
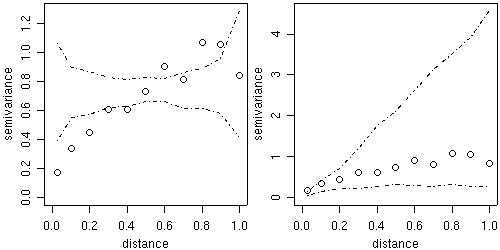
定向变异函数也可以通过函数variog使用参数direction和tolerance来计算。 例如,要使用默认容差角度(22.5度)计算60度方向的变差函数,命令为:
vario60 <- variog(s100, max.dist = 1, direction=pi/3)
plot(vario60)
title(main = expression(paste("directional, angle = ", 60 * degree)))
绘图结果显示在下图的左侧。
为了在四个方向快速计算,我们可以使用函数variog4,相应的图显示在下图的右侧。
vario.4 <- variog4(s100, max.dist = 1)
plot(vario.4, lwd=2)
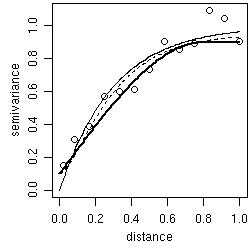
可以估计模型参数:
以下命令显示如何将变差函数模型添加到变量图。
plot(variog(s100, max.dist=1))
lines.variomodel(cov.model="exp", cov.pars=c(1,.3), nug=0, max.dist=1)
lines.variomodel(cov.model="mat", cov.pars=c(.85,.2), nug=0.1, kappa=1,max.dist=1, lty=2)
lines.variomodel(cov.model="sph", cov.pars=c(.8,.8), nug=0.1,max.dist=1, lwd=2)
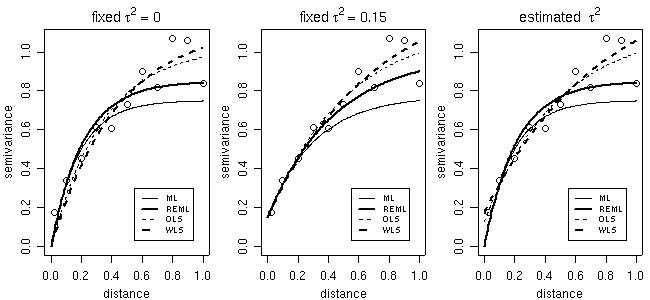
在参数估计函数variofit和likfit中,可以将nugget参数估计得出或设置为固定值(同样适用于平滑度,各向异性和变形参数)。 采取趋势考虑的选项也包括在内。 趋势可以被指定为给定协变量的坐标和/或线性函数的多项式函数。
下面的命令显示了通过不同方法配置的模型,其中包含固定/估计的nugget参数的选项。 这里未展示的特征包括趋势估计,各向异性,平滑度和Box-Cox变换参数。
# Fitting models with nugget fixed to zero
ml <- likfit(s100, ini = c(1,0.5), fix.nugget = T)
reml <- likfit(s100, ini = c(1,0.5), fix.nugget = T, method = "RML")
ols <- variofit(bin1, ini = c(1,0.5), fix.nugget = T, weights="equal")
wls <- variofit(bin1, ini = c(1,0.5), fix.nugget = T)
# Fitting models with a fixed value for the nugget
ml.fn <- likfit(s100, ini = c(1,0.5), fix.nugget = T, nugget = 0.15)
reml.fn <- likfit(s100, ini = c(1,0.5), fix.nugget = T, nugget = 0.15, method = "RML")
ols.fn <- variofit(bin1,ini = c(1,0.5), fix.nugget = T, nugget = 0.15, weights="equal")
wls.fn <- variofit(bin1, ini = c(1,0.5), fix.nugget = T, nugget = 0.15)
# Fitting models estimated nugget
ml.n <- likfit(s100, ini = c(1,0.5), nug = 0.5)
reml.n <- likfit(s100, ini = c(1,0.5), nug = 0.5, method = "RML")
ols.n <- variofit(bin1, ini = c(1,0.5), nugget=0.5, weights="equal")
wls.n <- variofit(bin1, ini = c(1,0.5), nugget=0.5)
# Now, plotting fitted models against empirical variogram
par(mfrow = c(1,3))
plot(bin1, main = expression(paste("fixed ", tau^2 == 0)))
lines(ml, max.dist = 1)
lines(reml, lwd = 2, max.dist = 1)
lines(ols, lty = 2, max.dist = 1)
lines(wls, lty = 2, lwd = 2, max.dist = 1)
legend(0.5, 0.3, legend=c("ML","REML","OLS","WLS"),lty=c(1,1,2,2),lwd=c(1,2,1,2), cex=0.7)
plot(bin1, main = expression(paste("fixed ", tau^2 == 0.15)))
lines(ml.fn, max.dist = 1)
lines(reml.fn, lwd = 2, max.dist = 1)
lines(ols.fn, lty = 2, max.dist = 1)
lines(wls.fn, lty = 2, lwd = 2, max.dist = 1)
legend(0.5, 0.3, legend=c("ML","REML","OLS","WLS"), lty=c(1,1,2,2), lwd=c(1,2,1,2), cex=0.7)
plot(bin1, main = expression(paste("estimated ", tau^2)))
lines(ml.n, max.dist = 1)
lines(reml.n, lwd = 2, max.dist = 1)
lines(ols.n, lty = 2, max.dist = 1)
lines(wls.n, lty =2, lwd = 2, max.dist = 1)
legend(0.5, 0.3, legend=c("ML","REML","OLS","WLS"), lty=c(1,1,2,2), lwd=c(1,2,1,2), cex=0.7)
par(par.ori)
已经编写了总结方法来总结生成的对象。例如,对于通过maximum likelihood拟合nugget估计的模型,输入:
ml.n
将产生输出:
likfit: estimated model parameters:
beta tausq sigmasq phi
0.7766 0.0000 0.7517 0.1827
likfit: maximised log-likelihood = -83.5696
获得更详细的摘要:
> summary(ml.n)
Summary of the parameter estimation
-----------------------------------
Estimation method: maximum likelihood
Parameters of the mean component (trend):
beta
0.7766
Parameters of the spatial component:
correlation function: exponential
(estimated) variance parameter sigmasq (partial sill) = 0.7517
(estimated) cor. fct. parameter phi (range parameter) = 0.1827
anisotropy parameters:
(fixed) anisotropy angle = 0 ( 0 degrees )
(fixed) anisotropy ratio = 1
Parameter of the error component:
(estimated) nugget = 0
Transformation parameter:
(fixed) Box-Cox parameter = 1 (no transformation)
Maximised Likelihood:
log.L n.params AIC BIC
-83.5696 4.0000 175.1391 185.5598
Call:
likfit(geodata = s100, ini.cov.pars = c(1, 0.5), nugget = 0.5)
通过仿真计算的两种变差函数envelopes如下图所示。
左侧的图表显示了基于跨位置的数据值排列envelope,即在没有空间相关性的假设下构建的envelopes。
右侧所示的envelope基于来自一组给定的模型参数的模拟,在该示例中,通过WLS变差函数拟合估计参数。 这个envelope显示了经验变异函数的变异性。
env.mc <- variog.mc.env(s100, obj.var=bin1)
env.model <- variog.model.env(s100, obj.var=bin1, model=wls)
par(mfrow=c(1,2))
plot(bin1, envelope=env.mc)
plot(bin1, envelope=env.model)
par(par.ori)
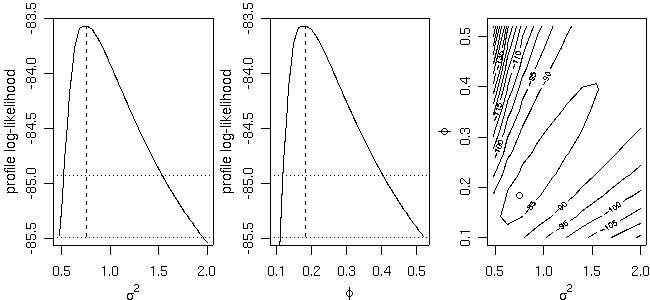
Profile likelihoods(1-D和2-D)由函数proflik计算。 在这里,我们展示了模型的协方差参数的profile likelihoods,不具有以前由likfit拟合的nugget效应。
WARNING:运行下一个命令将会消耗大量时间
prof <- proflik(ml, geodata = s100, sill.val = seq(0.48, 2, l=11),
range.val = seq(0.1, 0.52, l=11), uni.only = FALSE)
par(mfrow=c(1,3))
plot(prof, nlevels=16)
par(par.ori)
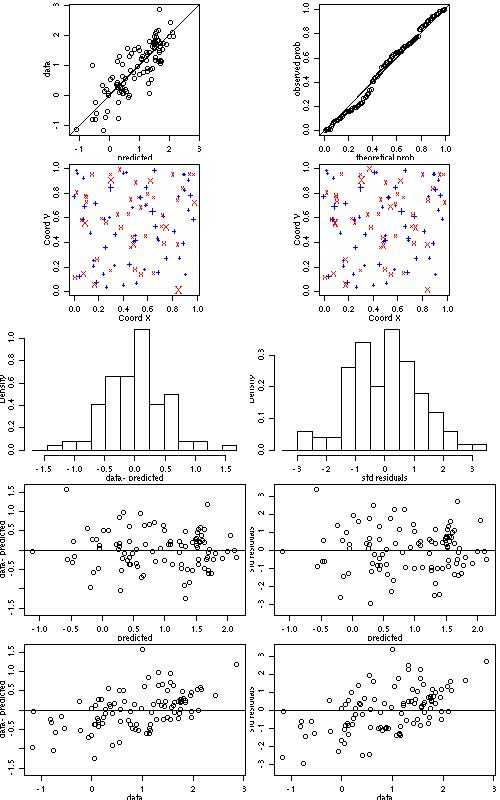
函数xvalid使用leaving-one-out策略或用户提供的一组互异位置执行交叉验证。 对于第一种策略,逐一删除数据点,并通过使用剩余数据进行克里金预测。下面的命令说明了通过最大似然和加权最小二乘拟合的模型的交叉验证。在前两个调用中,模型参数对于每个位置的预测保持相同。在接下来的两次调用中,每次从数据集中移除一个点时,将重新估计模型参数。 显示交叉验证结果的图形结果,其中leaving-one-out策略与参数的wls估计结合使用。
xv.ml <- xvalid(s100, model=ml)
xv.wls <- xvalid(s100, model=wls)
WARNING:运行下一个命令将会消耗大量时间
xvR.ml <- xvalid(s100, model=ml, reest=TRUE)
xvR.wls <- xvalid(s100, model=wls, reest=TRUE, variog.obj=bin1)
par(mfcol = c(5,2), mar=c(3,3,.5,.5), mgp=c(1.5,.7,0))
plot(xv.wls)
par(par.ori)
可以执行以下常规的地统计空间插值(克里金)选项:
o简单克里格
o普通克里金
o趋势(通用)克里金
o外部趋势克里金
还可执行Box-Cox转换(和结果的反向转换)和各向异性模型还有其他选项。
如果需要,可以从所得到的预测分布中获得模拟。
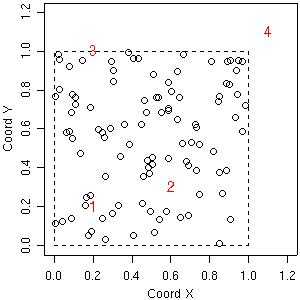
第一个例子计算下图中指出的、标记为1,2,3,4四个位置处的预测。
plot(s100$coords, xlim=c(0,1.2), ylim=c(0,1.2), xlab="Coord X", ylab="Coord Y")
loci <- matrix(c(0.2, 0.6, 0.2, 1.1, 0.2, 0.3, 1.0, 1.1), ncol=2)
text(loci, as.character(1:4), col="red")
polygon(x=c(0,1,1,0), y=c(0,0,1,1), lty=2)
使用nugget固定为零的加权最小二乘估计的参数执行普通克里金的命令是:
kc4 <- krige.conv(s100, locations = loci, krige = krige.control(obj.m = wls))
输出是包括预测值(kc4 $ predict)和克里格方差(kc4 $ krige.var)的列表。
第二个例子。 目标是在覆盖该区域的网格上执行预测并显示结果。 也使用普通克里金,命令是:
# defining the grid
pred.grid <- expand.grid(seq(0,1, l=51), seq(0,1, l=51))
# kriging calculations
kc <- krige.conv(s100, loc = pred.grid, krige = krige.control(obj.m = ml))
# displaying predicted values
image(kc, loc = pred.grid, col=gray(seq(1,0.1,l=30)), xlab="Coord X", ylab="Coord Y")

高斯模型的贝叶斯分析由函数krige.bayes实现。 它可以针对不同的“不确定度”执行,这意味着模型参数可以被视为是固定或随机的。
例如,考虑一个没有nugget的模型,包括平均值,基准和范围参数的不确定性。 上述四个位置的预测是通过键入如下命令来执行的:
WARNING:运行下一个命令将会消耗大量时间
bsp4 <- krige.bayes(s100, loc = loci, prior = prior.control(phi.discrete = seq(0,5,l=101), phi.prior="rec"), output=output.control(n.post=5000))
模型参数的后验分布直方图可以通过键入以下命令来绘制:
par(mfrow=c(1,3), mar=c(3,3,.5,.5), mgp=c(2,1,0))
hist(bsp4$posterior$sample$beta, main="", xlab=expression(beta), prob=T)
hist(bsp4$posterior$sample$sigmasq, main="", xlab=expression(sigma^2), prob=T)
hist(bsp4$posterior$sample$phi, main="", xlab=expression(phi), prob=T)
par(par.ori)
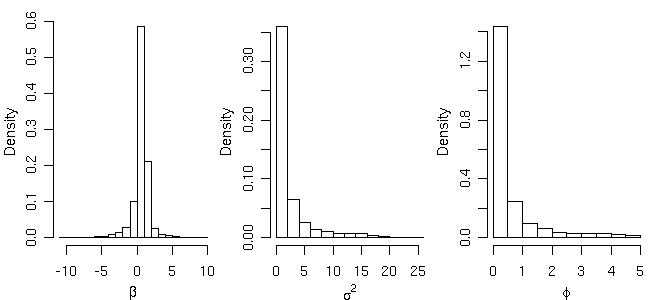
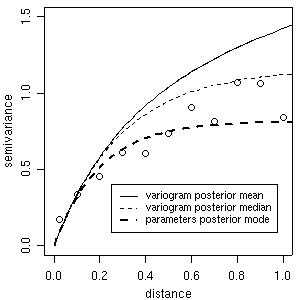
使用这些后验分布(平均值,中位数或模式)的汇总,我们可以根据经验变差函数检查“估计贝叶斯变异函数”,如下图所示。 请注意,也可以将这些估计与其他方法拟合的变异函数比较,如第3节中计算的。
plot(bin1, ylim = c(0,1.5))
lines(bsp4, max.dist = 1.2, summ = mean)
lines(bsp4, max.dist = 1.2, summ = median, lty = 2)
lines(bsp4, max.dist = 1.2, summ = "mode", post="par",lwd = 2, lty = 2)
legend(0.25, 0.4, legend = c("variogram posterior mean", "variogram posterior median", "parameters posterior mode"), lty = c(1,2,2), lwd = c(1,1,2), cex = 0.8)
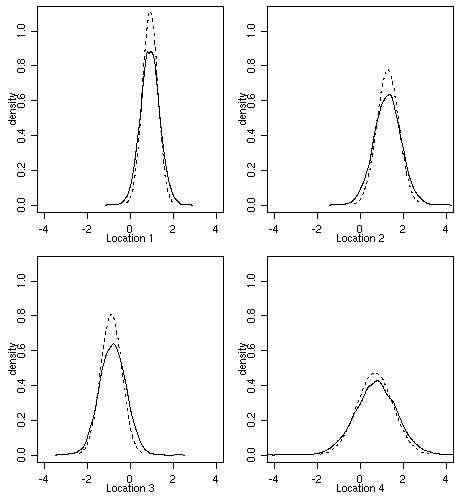
下图显示了四个选定位置的预测分布。
虚线显示了由第4节获得的OK结果给出的均值和方差的高斯分布。
实线对应于贝叶斯预测。 该图显示了使用预测分布的样本的密度估计结果。
par(mfrow=c(2,2), mar=c(3,3,.5,.5), mgp=c(1.5,.7,0))
for(i in 1:4){
kpx <- seq(kc4$pred[i] - 3*sqrt(kc4$krige.var[i]), kc4$pred[i] +3*sqrt(kc4$krige.var[i]), l=100)
kpy <- dnorm(kpx, mean=kc4$pred[i], sd=sqrt(kc4$krige.var[i]))
bp <- density(bsp4$predic$sim[i,])
rx <- range(c(kpx, bp$x))
ry <- range(c(kpy, bp$y))
plot(cbind(rx, ry), type="n", xlab=paste("Location", i), ylab="density", xlim=c(-4, 4), ylim=c(0,1.1))
lines(kpx, kpy, lty=2)
lines(bp)
}
par(par.ori)
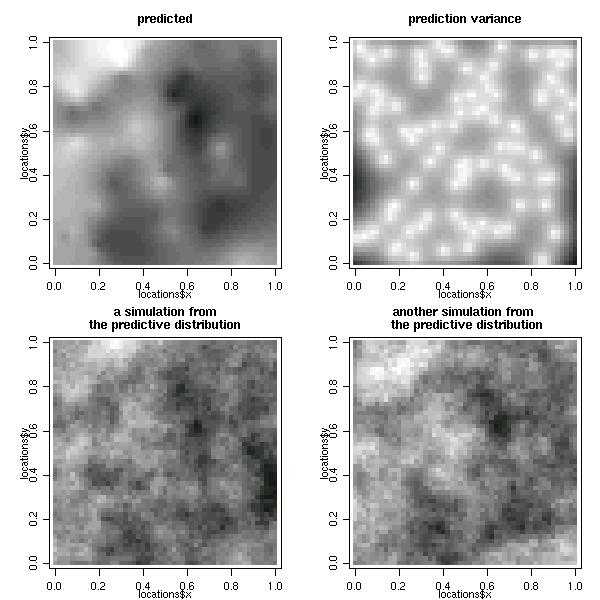
现在考虑在相同的模型假设下,从覆盖该区域的点的网格上的预测分布中获得模拟。 定义网格和执行贝叶斯预测的命令有:
pred.grid <- expand.grid(seq(0,1, l=31), seq(0,1, l=31))
WARNING:运行下一个命令将会消耗大量时间
bsp <- krige.bayes(s100, loc = pred.grid, prior = prior.control(phi.discrete = seq(0,5,l=51)),
output=output.control(n.predictive=2))
具有预测分布的总结和模拟的图形绘制如下。
par(mfrow=c(2,2))
image(bsp, loc = pred.grid, main = "predicted", col=gray(seq(1,0.1,l=30)))
image(bsp, val ="variance", loc = pred.grid,
main = "prediction variance", col=gray(seq(1,0.1,l=30)))
image(bsp, val = "simulation", number.col = 1, loc = pred.grid,
main = "a simulation from\nthe predictive distribution", col=gray(seq(1,0.1,l=30)))
image(bsp, val = "simulation", number.col = 2,loc = pred.grid,
main = "another simulation from \n the predictive distribution", col=gray(seq(1,0.1,l=30)))
par(par.ori)
注意:函数krige.bayes的更多示例在文件examples.krige.bayes.R中给出
函数grf在规则/不规则的位置集上生成高斯随机场的模拟。
其一些功能由下一个命令说明。
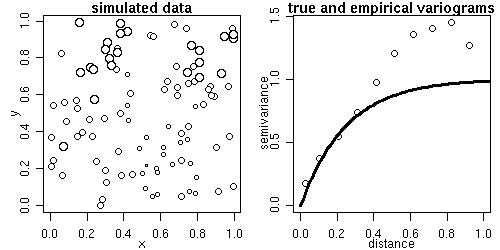
sim1 <- grf(100, cov.pars=c(1, .25))
points.geodata(sim1, main="simulated locations and values")
plot(sim1, max.dist=1, main="true and empirical variograms")
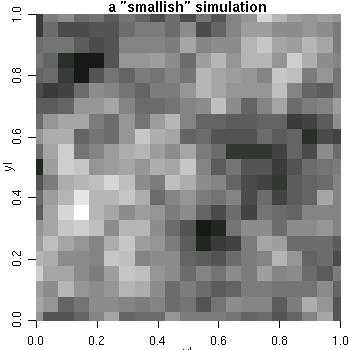
sim2 <- grf(441, grid="reg", cov.pars=c(1, .25))
image(sim2, main="a small-ish simulation", col=gray(seq(1, .1, l=30)))
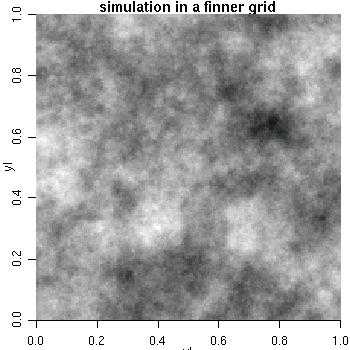
sim3 <- grf(40401, grid="reg", cov.pars=c(10, .2), met="circ")
image(sim3, main="simulation on a fine grid", col=gray(seq(1, .1, l=30)))
注意:我们建议使用RandomFields包来更全面地实现高斯随机场的模拟。
函数cite.geoR()显示有关如何在出版物中引用geoR的信息。
> cite.geoR()
To cite geoR in publications, use
RIBEIRO Jr., P.J. & DIGGLE, P.J. (2001) geoR: A package for
geostatistical analysis. R-NEWS, Vol 1, No 2, 15-18. ISSN 1609-3631.
Please cite geoR when using it for data analysis!
A BibTeX entry for LaTeX users is
@Article{,
title = {{geoR}: a package for geostatistical analysis},
author = {Ribeiro Jr., P.J. and Diggle, P.J.},
journal = {R-NEWS},
year = {2001},
volume = {1},
number = {2},
pages = {15--18},
issn = {1609-3631},
url = {http://cran.R-project.org/doc/Rnews}
}
网站维护者:Paulo J. Ribeiro Jr.(Paulo.Ribeiro@est.ufpr.br)
转载请注明出处:http://www.cnblogs.com/jingnianq/p/6606159.html
标签:引用 max 通用 1.2 需要 rac enter 不规则 bic
原文地址:http://www.cnblogs.com/jingnianq/p/6606159.html