标签:table stanford 排序 data lda 组成 细节 assm sam
本周做数据的预处理,本次有两个方向,
第一个是用pca算法进行预处理,进行降维
第二方向是通过k-mean算法进行处理,想法是利用聚类分析中 的K-means算法对训练集进行预处理(在第二次任务中有说明)
|
时间 |
内容 |
|
4.2-4.8 |
进行k-mean算法处理数据,完成上周遗留的bug,写读书笔记。 |
|
4.9-4.15 |
开始写毕业论文,继续编写毕设代码。 |
|
4.16-4.22 |
大致程序可以运行。 |
|
4.23-4.29 |
继续编写毕设程序,修改细节部分,完成毕业论文初稿。 |
|
4.30-4.29 |
完善毕业论文和毕业设计 |
|
4.30-5.6 |
完善毕业设计和论文 |
|
5.6-5.12 |
毕业论文定稿和准备答辩 |
主成分分析(Principal Components Analysis)的数学公式和原理已在上次的任务书中有详细描述,这里为伪代码描述
第一步:求均值。求平均值,然后对于所有的样例,都减去对应的均值
第二步:求特征协方差矩阵
第三步:求协方差的特征值和特征向量
第四步:将特征值按照从大到小的顺序排序,选择其中最大的k个,然后将其对应的k个特征向量分别作为列向量组成特征向量矩阵.
第五步:将样本点投影到选取的特征向量上。 假设样例数为m,特征数为n,减去均值后的样本矩阵为DataAdjust(m*n),协方差矩阵是n*n,选取的k个特征向量组成的矩阵为EigenVectors(n*k).那么投影后的数据FinalData为: FinalData(m*k) = DataAdjust(m*n) * EigenVectors(n*k)。
K-mean算法描述
选择K个点作为初始质心
repeat
将每个点指派到最近的质心,形成K个簇
重新计算每个簇的质心
until 簇不发生变化或达到最大迭代次数
4.你本周实现代码,贴图核心代码,并注释。
pca主要代码
def pca(dataMat, K=65535): # dataMat是原始数据,一个矩阵,K是要降到的维数
dataMat = mat(dataMat)
# print(dataMat)
meanVals =
mean(dataMat, axis=0) # 第一步:求均值
meanRemoved =
dataMat - meanVals # 减去对应的均值
#
print(meanRemoved)
covMat =
cov(meanRemoved, rowvar=0) # 第二步,求特征协方差矩阵
# print(covMat)
eigVals, eigVects = linalg.eig(mat(covMat)) # 第三步,求特征值和特征向量
#
print(eigVects,eigVals)
eigValInd =
argsort(eigVals) # 第四步,将特征值按照从小到大的顺序排序
eigValInd =
eigValInd[: -(K + 1): -1] # 选择其中最大的K个
redEigVects =
eigVects[:, eigValInd] # 然后将其对应的k个特征向量分别作为列向量组成特征向量矩阵.
lowDDataMat =
meanRemoved * redEigVects # 第五步,将样本点投影到选取的特征向量上,得到降维后的数据
reconMat = (lowDDataMat * redEigVects.T) + meanVals # 还原数据
# # contribution =
self.calc_single_contribute(eigVals, eigValInd)
# 计算单维贡献度,总贡献度为其和
return lowDDataMat, reconMat
k-mean主要代码
def kmeans(dataSet, k):
numSamples = dataSet.shape[0]
clusterAssment = np.mat(np.zeros((numSamples, 2)))
clusterChanged = True
## step 1: init
centroids
centroids =
initCentroids(dataSet, k)
while clusterChanged:
clusterChanged = False
## for each sample
for i in range(numSamples):
minDist = 100000.0 # 与最近族群距离
minIndex = 0 # 所属族
## for each centroid
## step 2: find the centroid
who is closest
for j in range(k):
distance =
euclDistance(centroids[j, :], dataSet[i, :])
print(distance)
if distance < minDist: # 更新最小距离,所属族
minDist = distance
minIndex = j
## step 3: update its cluster
if clusterAssment[i, 0] != minIndex: # 所属族群有变化
clusterChanged = True
clusterAssment[i, :] = minIndex, minDist ** 2 # 族群索引号,距离
## step 4: update
centroids
for j in range(k):
test1 = clusterAssment[:, 0] # 获取所属族群
test2 = clusterAssment[:, 0].A # 转换为数组
test3 = clusterAssment[:, 0].A == j # 判断是否属于族群J
test4 =
np.nonzero(test3) # 属于族群J的索引值
test5 = test4[0]
test6 = dataSet[test5]
pointsInCluster =
dataSet[np.nonzero(clusterAssment[:, 0].A == j)[0]]
# pointsInCluster =
dataSet[nonzero(clusterAssment[:, 0].A == j)[0]]
centroids[j, :] = np.mean(pointsInCluster, axis=0) # 所有族群元素特征值求平均
print(‘Congratulations, cluster complete!‘)
return centroids, clusterAssment
5.你本周实现效果截图。
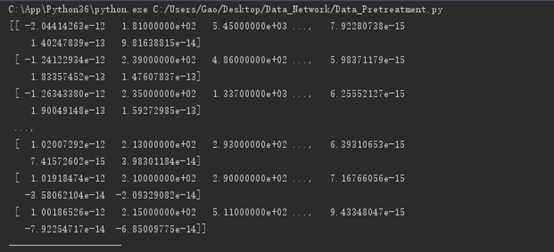
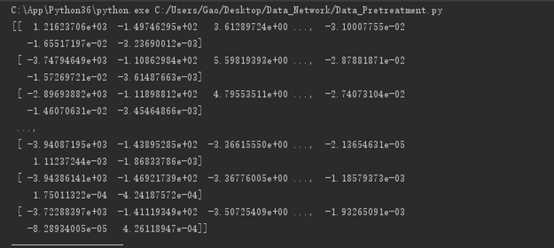
本次实验使用numpy包。显示数据自动省略,上图为经过pca算法后还原数据,下图为降维数据
6.你本周小结
本周主要是pca算法,后发现k-mean算法或许会更好一些,本周已完成k-mean算法,下周进行调试,和修改
在本周编写过程中,数据量有些大,pc测试时,效率很低,下周想使用数据库,将数据放入,看是否能够提高io效率
下周要对算法中对特征值选取的方法进行改进
7.本周参考
标签:table stanford 排序 data lda 组成 细节 assm sam
原文地址:http://www.cnblogs.com/G-Domain/p/6657513.html