标签:transform mit images standards 取数 方式 标记 性能分析 解决
逻辑斯蒂回归分类器
打开ipython网页解释器
#导入模块 import pandas as pd import numpy as np
#创建特征列表表头 column_names = [‘Sample code number‘,‘Clump Thickness‘,‘Uniformity of Cell Size‘,‘Uniformity of Cell Shape‘,‘Marginal Adhesion‘,‘Single Epithelial Cell Size‘,‘Bare Nuclei‘,‘Bland Chromatin‘,‘Normal Nucleoli‘,‘Mitoses‘,‘Class‘] #使用pandas.read_csv函数从网上读取数据集 data = pd.read_csv(‘https://archive.ics.uci.edu/ml/machine-learning-databases/breast-cancer-wisconsin/breast-cancer-wisconsin.data‘,names=column_names) #将?替换为标准缺失值表示 data = data.replace(to_replace=‘?‘,value = np.nan) #丢弃带有缺失值的数据(只要有一个维度有缺失便丢弃) data = data.dropna(how=‘any‘) #查看data的数据量和维度 data.shape
维度打印信息为:

有683条数据,维度为11
如果用mac环境,在读取网络上数据集时候可能会报错,解决方案,打开Finder,找到python解释器,点击安装即可
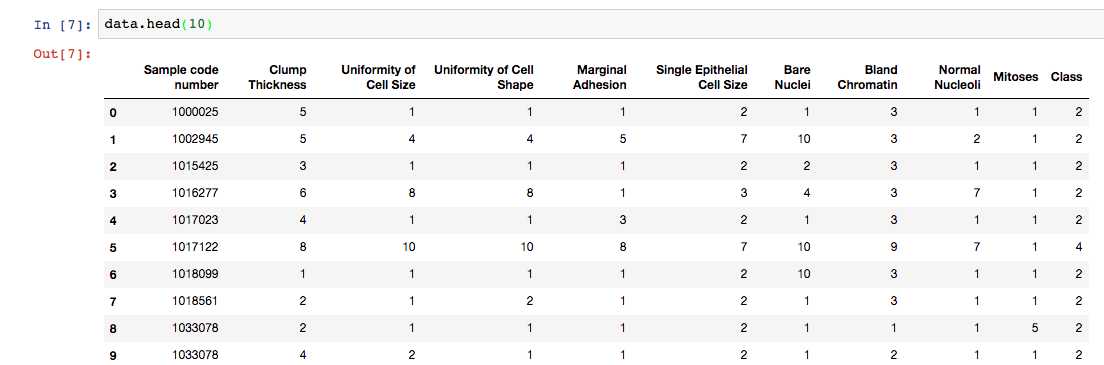
处理后数据集如下:
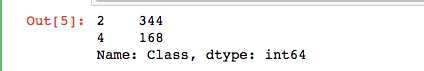
由于原始数据没有提供对应的测试样本用于评估模型性能,这里对带标记的数据进行分割,25%作为测试集,其余作为训练集
#使用sklearn.cross_validation里的train_test_split模块分割数据集 from sklearn.cross_validation import train_test_split #随机采样25%的数据用于测试,剩下的75%用于构建训练集 X_train,X_test,y_train,y_test = train_test_split(data[column_names[1:10]],data[column_names[10]],test_size = 0.25,random_state = 33) #查看训练样本的数量和类别分布 y_train.value_counts()
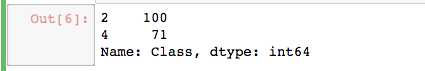
#查看测试样本的数量和类别分布 y_test.value_counts()
#从sklearn.preprocessing导入StandardScaler from sklearn.preprocessing import StandardScaler #从sklearn.linear_model导入LogisticRegression(逻辑斯蒂回归) from sklearn.linear_model import LogisticRegression #从sklearn.linear_model导入SGDClassifier(随机梯度参数) from sklearn.linear_model import SGDClassifier
#标准化数据,保证每个维度的特征数据方差为1,均值为,使得预测结果不会被某些过大的特征值而主导(在机器学习训练之前, 先对数据预先处理一下, 取值跨度大的特征数据,
我们浓缩一下, 跨度小的括展一下, 使得他们的跨度尽量统一.) ss = StandardScaler() X_train = ss.fit_transform(X_train) X_test = ss.transform(X_test)
#初始化两种模型 lr = LogisticRegression() sgdc = SGDClassifier()
#调用逻辑斯蒂回归,使用fit函数训练模型参数 lr.fit(X_train,y_train)
#使用训练好的模型lr对x_test进行预测,结果储存在变量lr_y_predict中 lr_y_predict = lr.predict(X_test)
#调用随机梯度的fit函数训练模型 sgdc.fit(X_train,y_train)
#使用训练好的模型sgdc对X_test进行预测,结果储存在变量sgdc_y_predict中 sgdc_y_predict = sgdc.predict(X_test)
下面查看两种模型的预测结果

#从sklearn.metrics导入classification_report from sklearn.metrics import classification_report #使用逻辑斯蒂回归模型自带的评分函数score获得模型在测试集上的准确性结果 print(‘Accuracy of LR Classifier:‘,lr.score(X_test,y_test)) #使用classification_report模块获得逻辑斯蒂模型其他三个指标的结果(召回率,精确率,调和平均数) print(classification_report(y_test,lr_y_predict,target_names=[‘Benign‘,‘Malignant‘]))

#使用随机梯度下降模型自带的评分函数score获得模型在测试集上的准确性结果 print(‘Accuarcy of SGD Classifier:‘,sgdc.score(X_test,y_test)) ##使用classification_report模块获得随机梯度下降模型其他三个指标的结果 print(classification_report(y_test,sgdc_y_predict,target_names=[‘Benign‘,‘Malignant‘]))
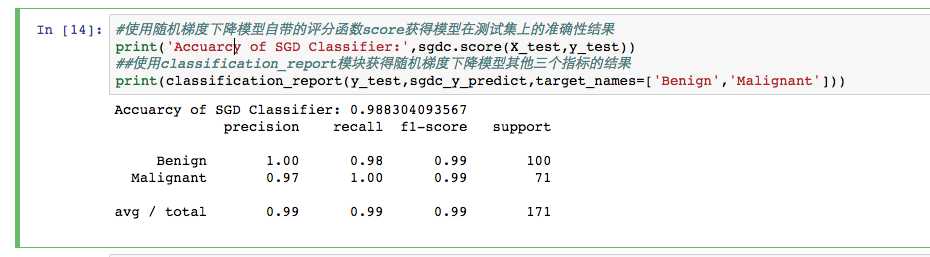
结论:通过比较,逻辑斯蒂模型比随机梯度下降模型在测试集上表现有更高的准确性,因为逻辑斯蒂采用解析的方式精确计算模型参数,而随机梯度下降采用估计值
特点分析:逻辑斯蒂对参数的计算采用精确解析的方法,计算时间长但是模型性能高,随机梯度下降采用随机梯度上升算法估计模型参数,计算时间短但产出的模型性能略低,一般而言,对于训练数据规模在10万量级以上的数据,考虑到时间的耗用,推荐使用随机梯度算法
标签:transform mit images standards 取数 方式 标记 性能分析 解决
原文地址:http://www.cnblogs.com/chenice/p/7202045.html