标签:pat htm ksh row ack img images 用户 深度
官网说明书:http://genome.ucsc.edu/goldenpath/help/hgTracksHelp.html
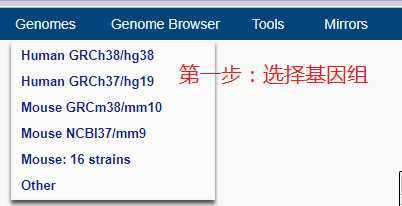
1.genome brower的作用
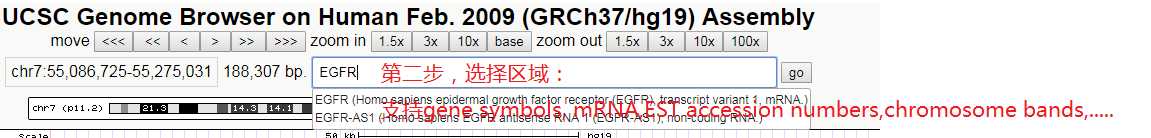
a,展示任何尺度的基因组片段。比如,想看某条染色体的某个区域的序列。
b,展示某些位置的annotation tracks。比如,你得到一些区域的深度,你想查看下这些区域的深度在染色体上怎么分布的,他们是否有一致性。
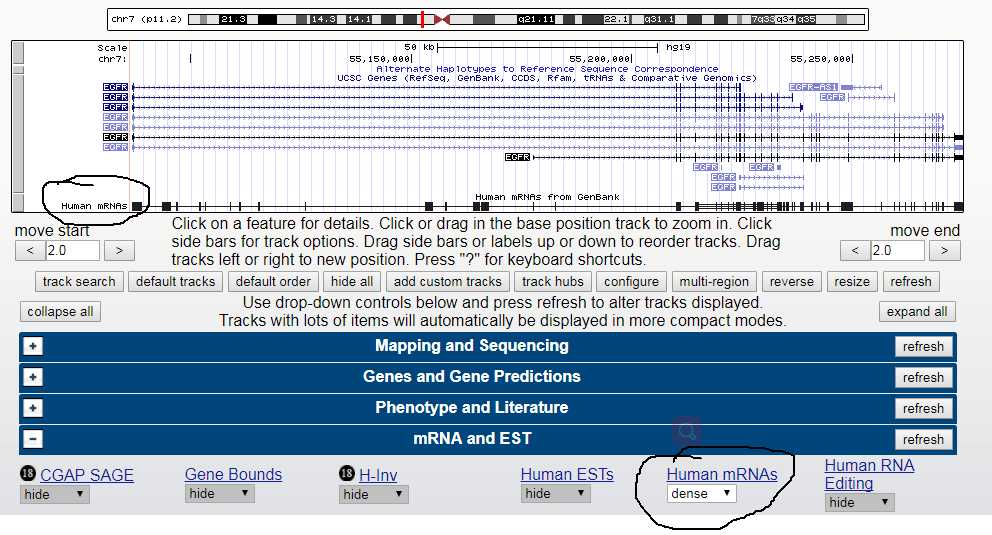
annotation tracks展示在基因组坐标的下方。
ucsc自身带有一些annotaton tracks,一半来自已出版的文章,另一半来自其合作者。用户也可以自定义annotation tracks,这边会涉及到tracks的文件格式。
2.genome brower的开始


标签:pat htm ksh row ack img images 用户 深度
原文地址:http://www.cnblogs.com/timeisbiggestboss/p/7742886.html