标签:smr back idg ons img sub 格式 AC seq
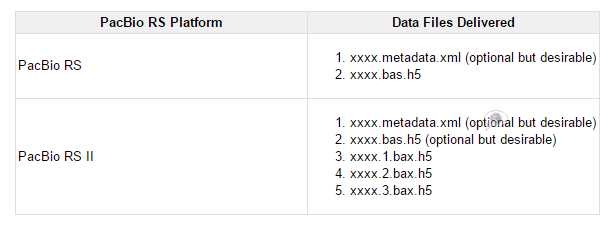
2.格式转换
(1) bas.h5 -> ccs
source /share/nas2/genome/biosoft/smrtanalysis/2.3.0/smrtanalysis/current/etc/setup.sh
bash5tools.py --readType subreads --outType fasta /path/to/bas.h5
(2) bax.h5 -> ccs
需要各个文件进行转换并进行合并
标签:smr back idg ons img sub 格式 AC seq
原文地址:https://www.cnblogs.com/zwj-pot/p/8861712.html