标签:library 输入数据 color 注意 exp format 文件 display time
转自:http://www.omicshare.com/forum/thread-741-1-1.html
范例文件(txt)是一个20个样本,30个基因的表达量表格矩阵。每一行是1个基因,每一列对应1个样本。

这是一个典型的数据框文件。现在我要计算两两样本间的表达量的相关系数,并且对相关系数的结果绘制热图,该怎么做呢?只要两步:
(1)计算相关系数;
在R语言里面,相关系数的命令是 cor。这个命令是可以计算两个向量的相关系数。但你如果输入数据是数据框的,而且cor命令自动计算所有列(向量)的两两相关系数(范例文件列方向正好是样本)。
(2)绘制热图
绘制热图可以使用pheatmap这个命令。
代码如下:
############
library(pheatmap) #加载pheatmap 包;
data=read.table("exp_top30.original.txt",header=T,row.names=1,sep="\t")
matrix=cor(data) #计算相关系数;
write.table(matrix,"coefficient_matrix.txt",sep="\t") #将相关系数计算结果输出存储到你的电脑里,存储为1个txt文件;
pheatmap(matrix,cluster_rows=F,cluster_cols=F,display_numbers=T) # 行和列都不聚类,并且在热图中显示数值;
#####################
效果图:
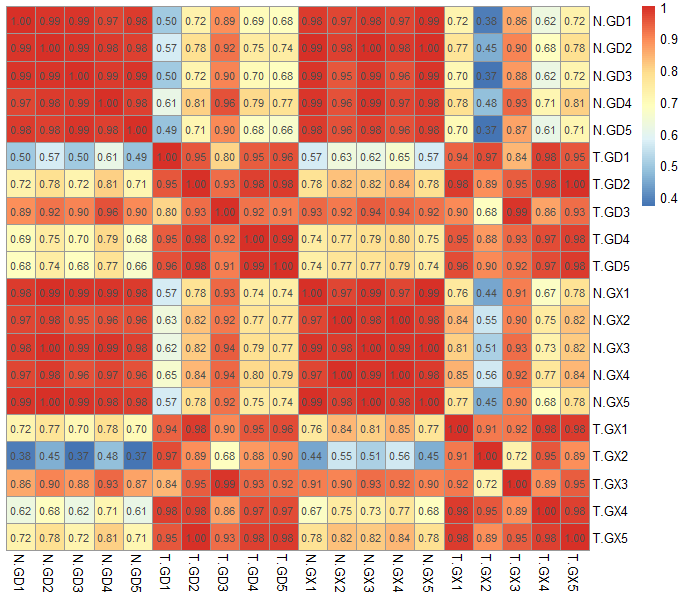
如果要计算每一行的相关系数(这里行方向是基因,即行间相关系数就是基因间的相关系数),则需要对矩阵做个转置,即行列对调,使用t()命令即可,
代码如下:
###################
library(pheatmap) #加载pheatmap 包;
data=read.table("exp_top30.original.txt",header=T,row.names=1,sep="\t")
data=t(data) # 对数据做转置;
matrix=cor(data) #计算相关系数;
write.table(matrix,"coefficient_matrix.txt",sep="\t") #将相关系数计算结果输出存储到你的电脑里,存储为1个txt文件;
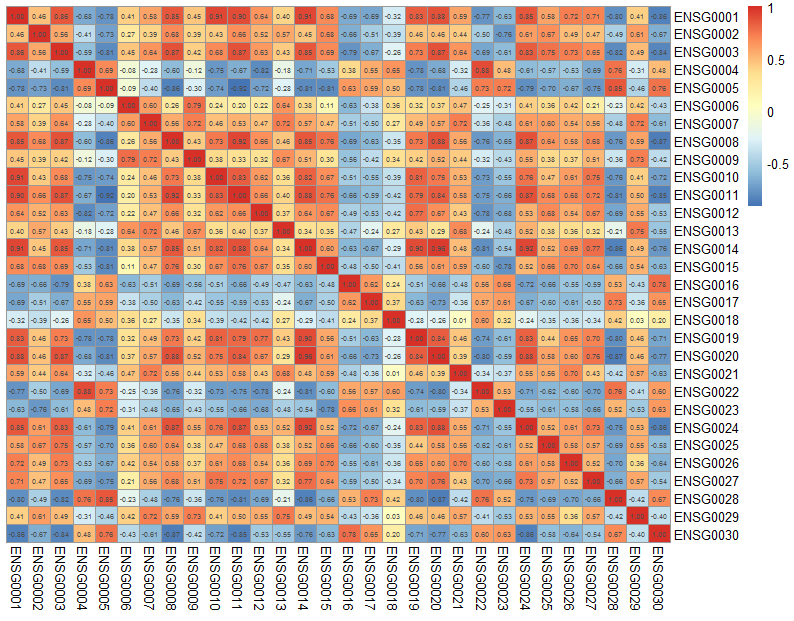
pheatmap(matrix,cluster_rows=F,cluster_cols=F,display_numbers=T,fontsize_number=4,number_format = "%.2f") # 注意,由于格子比较多,所以用fontsize_number定义了格子中数字的字体大小, number_format 可以控制有效小数的位数,这里是保留两位小数;
#################################
效果图如下:
标签:library 输入数据 color 注意 exp format 文件 display time
原文地址:https://www.cnblogs.com/djx571/p/9216849.html