标签:copy 数据 splay detail lan col rod err cat
一、什么是高斯混合模型(GMM)
高斯混合模型(Gaussian Mixed Model)指的是多个高斯分布函数的线性组合,通常用于解决同一集合下的数据包含多个不同的分布的情况,如利用GMM来解决分类情况,
如下图,在我们看来明显分成两个聚类。这两个聚类中的点分别通过两个不同的正态分布随机生成而来。但是如果没有GMM,那么只能用一个的二维高斯分布来描述图中的数据。这显然不太合理,毕竟肉眼一看就觉得应该把它们分成两类
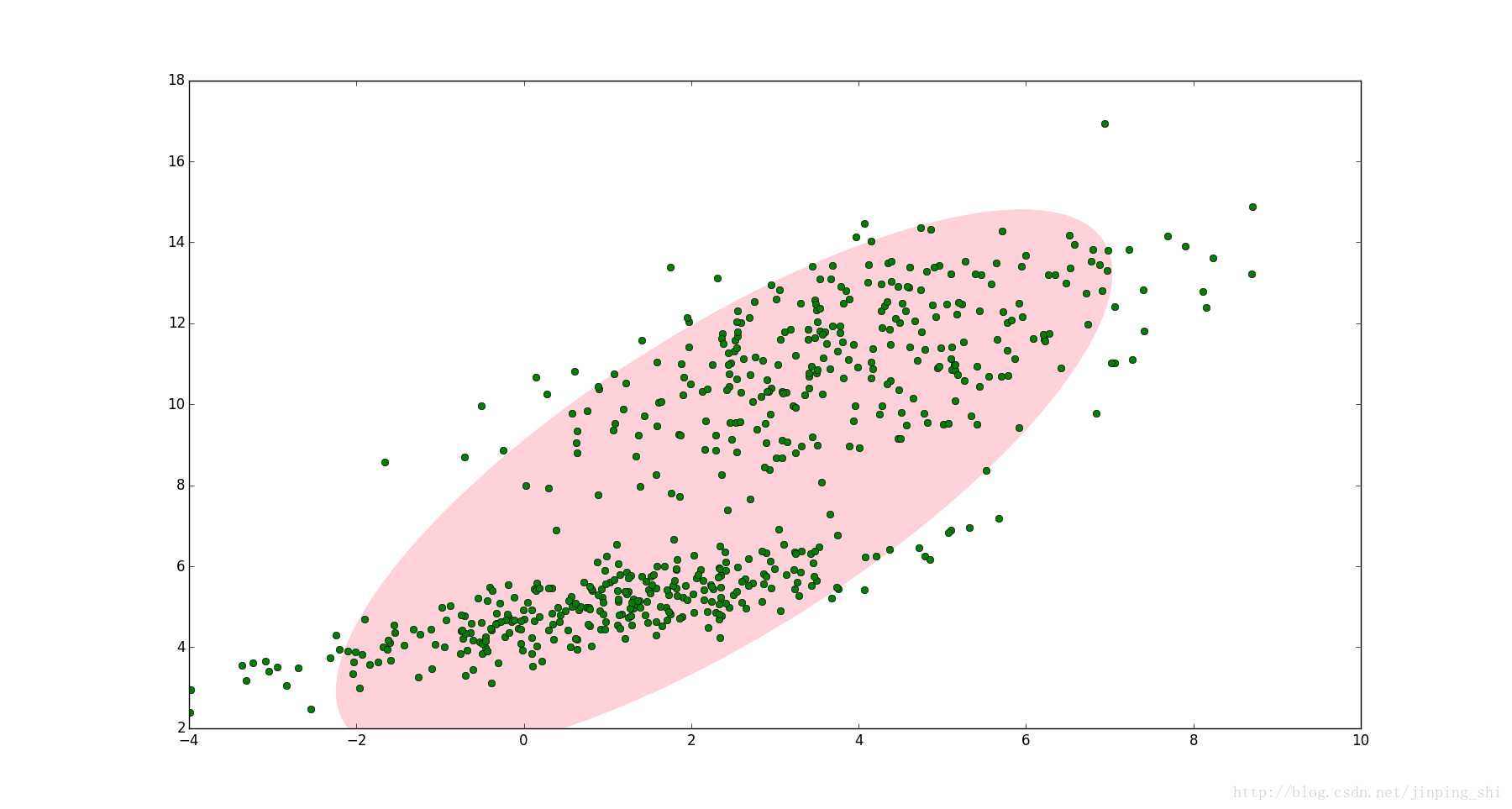
这时可以使用两个二维高斯分布来描述图中的数据,将两个二维高斯分布\(N(\mu_1,\sum_1),N(\mu_2,\sum_2)\)做线性组合,用线性组合后的分布来描述整个集合中的数据。这就是高斯混合模型(GMM)
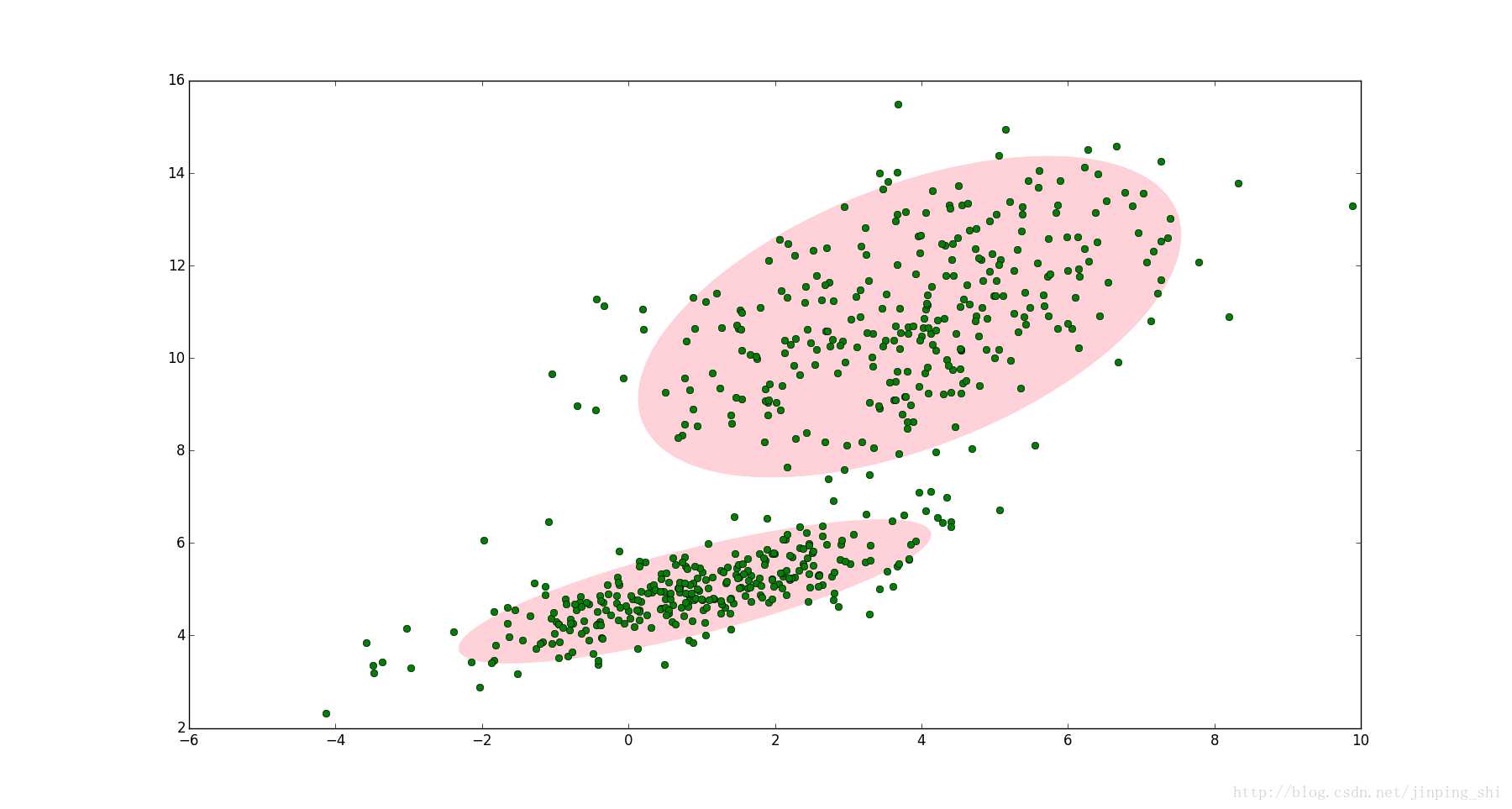
二、GMM原理
设有随机变量\(X\),则混合高斯模型可以这样表示:
\(p(x) = \sum_{k=1}^{K}\pi_kN(x|\mu_k,\sum_k)\)
其中\(N(x|\mu_k,\sum_k)\)称为混合模型中的第k个分量(component)。如前面图中的例子,有两个聚类,可以用两个二维高斯分布来表示,那么分量数K=2,\(\pi_k\)是混合系数(mixture coefficient),且满足:
\( \sum_{k=1}^{K}\pi_k,0\leq\pi_k\leq 1 \),其中\(\pi_k\)相当于每个高斯分布的权重
GMM用于聚类时,假设数据服从混合高斯分布(Mixture Gaussian Distribution),例如上图的例子,很明显有两个聚类,可以定义K=2,那么对应的GMM形式如下:
\( p(x) = \pi_1N(x|\mu_1,\sum_1) + \pi_2N(x|\mu_2,\sum_2) \)
上式中未知参数有六个:\((\pi_1,\mu_1,\sum_1, \pi_2,\mu_2,\sum_2)\),GMM聚类时分为两步,第一步是随机地在这K个分量中选一个,每个分量被选中的概率即为混合系数\(\pi_k\),可以设定\(\pi_1=\pi_2=0.5\),表示每个分量被选中的概率是0.5,即从中抽出一个点,这个点属于第一类的概率和第二类的概率各占一半。但实际应用中事先指定\(\pi_k\)的值是很笨的做法,当问题一般化后,会出现一个问题:当从图中的集合随机选取一个点,怎么知道这个点是来自\(N(x|\mu_1,\sum_1)\)还是\(N(x|\mu_2,\sum_2)\)呢?
换言之怎么根据数据自动确定\(\pi_1,\pi_2\)的值?这就是GMM参数估计的问题。要解决这个问题,可以使用EM算法。通过EM算法,我们可以迭代计算出GMM中的参数:\(\pi_k,\mu_k,\sum_k\)
我们的想法是,找到这样一组参数,它所确定的概率分布生成这些给定的数据点的概率最大,而这个概率实际上就等于:\(\prod_{i=1}^{N}p(x_i)\),我们把这个乘积称作似然函数 (Likelihood Function)。通常单个点的概率都很小,许多很小的数字相乘起来在计算机里很容易造成浮点数下溢,因此我们通常会对其取对数,把乘积变成加和:\(\sum_{i=1}^{N}logp(x_i)\),得到 log-likelihood function 。接下来我们只要将这个函数最大化(通常的做法是求导并令导数等于零,然后解方程),亦即找到这样一组参数值,它让似然函数取得最大值,我们就认为这是最合适的参数,这样就完成了参数估计的过程。下面让我们来看一看 GMM 的 log-likelihood function :
\(\sum_{i=1}^{N}log\{\sum_{i=1}^{K}\pi_kN(x_i|\mu_k,\sum_k)\}\)
由于上式太复杂,没法直接用求导求得最大值。为了解决这个问题,我们采取之前从 GMM 中随机选点的办法:分成两步
1.估计每个数据由每个 Component 生成的概率(并不是每个 Component 被选中的概率):对于每个数据\(x_i\)来说,它由第\(k\)个 Component 生成的概率为:
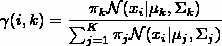
由于式子里的\(\mu_k,\sum_k\)也是需要我们估计的值,我们采用迭代法,在计算\(\gamma(i,k)\)的时候我们假定\(\mu_k,\sum_k\)均已知,我们将取上一次迭代所得的值(或者初始值)
2.估计每个Component 的参数:现在我们假设上一步中得到的\(\gamma(i,k)\)就是正确的"数据\(x_i\)由Component \(k\)生成的概率",亦可以当做该Component 在生成这个数据上所做的贡献,或者说,我们可以看作\(x_i\)这个数据其中有\(\gamma(i,k)x_i\)这部分是由Component \(k\)所生成的,集中考虑所有的数据点,现在实际上可以看作Component 生成了\( \gamma(1,k)x_1,...,\gamma(N,k)x_N \)这些点,由于每个 Component 都是一个标准的 Gaussian 分布,可以很容易求出最大似然所对应的参数值:
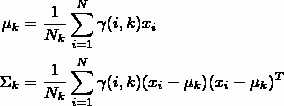
其中\(N_k = \sum_{i=1}^{N}\gamma(i,k)\),并且\(\pi_k\)也顺理成章地可以估计\(N_k/N\)
3.重复迭代前面两步,直到似然函数的值收敛为止
三、代码实例
参考:https://blog.csdn.net/u014157632/article/details/65442165
‘‘‘
此示例程序随机从4个高斯模型中生成500个2维数据,真实参数:
混合项w=[0.1,0.2,0.3,0.4]
均值u=[[5,35],[30,40],[20,20],[45,15]]
协方差矩阵∑=[[30,0],[0,30]]
然后以这些数据作为观测数据,根据EM算法来估计以上参数
此程序未估计协方差矩阵
‘‘‘
import math
import copy
import numpy as np
import matplotlib.pyplot as plt
from mpl_toolkits.mplot3d import Axes3D
iter_num = 1000
N = 500
k = 4
probility = np.zeros(N)
u1 = [5, 35]
u2 = [30, 40]
u3 = [20, 20]
u4 = [45, 15]
sigma = np.matrix([[30, 0], [0,30]])
alpha = [0.1, 0.2, 0.3, 0.4]
#生成随机数据,4个高斯模型
def generate_data(sigma, N, mu1, mu2, mu3, mu4, alpha):
global X#可观测数据集
X = np.zeros((N, 2))#X:2维数据,N个样本
X = np.matrix(X)
global mu#随机初始化mu1,mu2,mu3,mu4
mu = np.random.random((4, 2))
mu = np.matrix(mu)
global excep#期望:第i个样本属于第j个模型的概率的期望
excep = np.zeros((N, 4))
global alpha_#初始化混合项系数
alpha_ = [0.25, 0.25, 0.25, 0.25]
#np.random.multivariate_normal():用于根据实际情况生成一个多元正态分布矩阵
for i in range(N):
if np.random.random(1) < 0.1:
X[i, :] = np.random.multivariate_normal(mu1, sigma, 1)
elif 0.1 <= np.random.random(1) < 0.3:
X[i, :] = np.random.multivariate_normal(mu2, sigma, 1)
elif 0.3 <= np.random.random(1) < 0.6:
X[i, :] = np.random.multivariate_normal(mu3, sigma, 1)
else:
X[i, :] = np.random.multivariate_normal(mu4, sigma, 1)
print(‘可观测数据:\n‘, X)
print(‘初始化的mu1, mu2, mu3, mu4:‘, mu)
def e_step(sigma, k, N):
global X
global mu
global excep
global alpha_
for i in range(N):
denom = 0
for j in range(0, k):
denom += alpha_[j] * math.exp(-0.5 * (X[i,:]-mu[j,:])*sigma.I*np.transpose(X[i,:]-mu[j,:]))/np.sqrt(np.linalg.det(sigma))#分母
for j in range(0, k):
numer = math.exp(-0.5 * (X[i,:]-mu[j,:])*sigma.I*np.transpose(X[i,:]-mu[j,:]))/np.sqrt(np.linalg.det(sigma))#分子
excep[i, j] = alpha_[j] * numer / denom#求期望
print(‘隐藏变量:\n‘, excep)
def m_step(k, N):
global excep
global X
global alpha_
for j in range(0, k):
denom = 0#分母
numer = 0#分子
for i in range(N):
numer += excep[i, j] * X[i, :]
denom += excep[i, j]
mu[j, :] = numer / denom #求均值
alpha_[j] = denom / N#求混合项系数
generate_data(sigma, N, u1, u2, u3, u4, alpha)
#迭代计算
for i in range(iter_num):
err = 0#均值误差
err_alpha = 0#混合系数误差
Old_mu = copy.deepcopy(mu)
Old_alpha = copy.deepcopy(alpha_)
e_step(sigma, k, N)#E 步
m_step(k, N)#M 步
print("迭代次数:", i+1)
print("估计的均值:", mu)
print("估计的混合项系数:", alpha_)
for z in range(k):
err += (abs(Old_mu[z, 0] - mu[z, 0]) + abs(Old_mu[z, 1] - mu[z, 1]))#计算误差
err_alpha += abs(Old_alpha[z] - alpha_[z])
if (err <= 0.001) and (err_alpha < 0.001):#达到精度退出迭代
print(err, err_alpha)
break
#可视化结果,画生成的原始数据
plt.subplot(221)
plt.scatter(X[:,0].tolist(), X[:,1].tolist(),c=‘b‘, s=25, alpha=0.4, marker=‘o‘)
plt.title(‘random generated data‘)
#画分类好的数据
plt.subplot(222)
plt.title(‘classified data through EM‘)
order = np.zeros(N)
color = [‘b‘, ‘r‘, ‘k‘, ‘y‘]
for i in range(N):
for j in range(k):
if excep[i, j] == max(excep[i, :]):
order[i] = j#选出X[i, :]属于第几个高斯模型
probility[i] += alpha_[int(order[i])] * math.exp(-0.5 * (X[i,:]-mu[j,:])*sigma.I*np.transpose(X[i,:]-mu[j,:]))/(np.sqrt(np.linalg.det(sigma))*2*np.pi)#计算混合高斯分布
plt.scatter(X[i, 0], X[i, 1], c=color[int(order[i])], s=25, alpha=0.4, marker=‘o‘)#绘制分类后的散点图
#绘制三维图像
ax = plt.subplot(223, projection=‘3d‘)
plt.title(‘3d view‘)
for i in range(N):
ax.scatter(X[i,0], X[i, 1], probility[i], c=color[int(order[i])])
plt.show()
标签:copy 数据 splay detail lan col rod err cat
原文地址:https://www.cnblogs.com/always-fight/p/9327418.html