标签:特点 数据集 display 额外 png 不同 效果 实现 内存占用
最近看了一篇关于NGS(下一代测序)、ASAM的论文。名字为《AMAS: Optimizing the Partition and Filtration of Adaptive Seeds to Speed up Read Mapping》。这篇论文介绍了一种可以提高映射速度的测序技术——AMAS。总体上来说这个技术的映射速度、内存占用、稳定性都是最优的。ASAM测序方法使用的核心是与GEM(The Genome Multitool)相同的自适应种子。之后又介绍了与其他主流测序算法的对比。最后展示了在不同参数下,自身算法的效果。
本文只是学习随笔,简单介绍,如想深入学习请仔细研究原文。
读取映射是NGS数据分析中的关键任务。现在比较主流的读取映射器包括:SOAP 2, mrsFAST ,mrFAST , RazerS 3, GEM , Masai,Hobbes 1, 2。而ASMA与其他技术不同在于以下几个方面:
1. 当当前种子的匹配数下降到预定频率阈值“F”以下时,将其位置添加到候选空间然后启动一个新种子,直到获得所需数量的种子或达到读数结束。对于100k读取的整个模拟数据集,候选位置的总数减少了6.61倍
2. AMAS在索引步骤为每个参考基因组预先计算一次自适应种子,并将其信息存储在基数树中,用于每个未来的映射任务
3. 读取的自适应分区中的最后一个种子可能太短,其频率可能超过其他种子的频率以及阈值“F”。这些位置的大部分是误报,但是去除它们可能会导致映射灵敏度的丢失。ASMA只过滤掉那些匹配数量远远高于预期频率的种子,这种过滤步骤只影响少量的读数。但是从结果上看显著地减少了50%以上总候选空间。
4. ASMA将候选位置存储在二叉搜索树中,允许对其快速排序并识别多个种子报告。在映射步骤期间避免重复自适应种子的计算以减少映射时间。
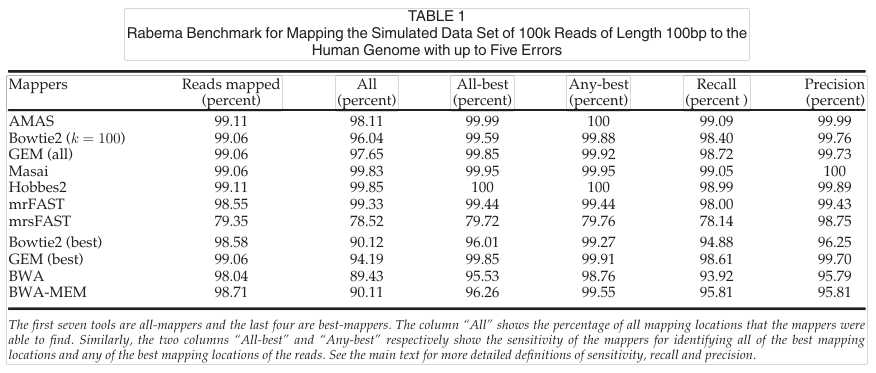
表1为100k,长度为100bp读数下,各个算法的效果。AMAS中引入的最大种子长度的附加约束保证了所有映射位置的完全灵敏度(100%)。GEM在运行时间和灵敏度方面都优于其他映射器,展示了自适应种子的优势。
表2为在1000基因组计划的真实数据集上运行映射器,单线程和八线程模式下ASMA都是很优秀的。这清楚地显示了AMAS中实现的优化分区和过滤方法的好处。
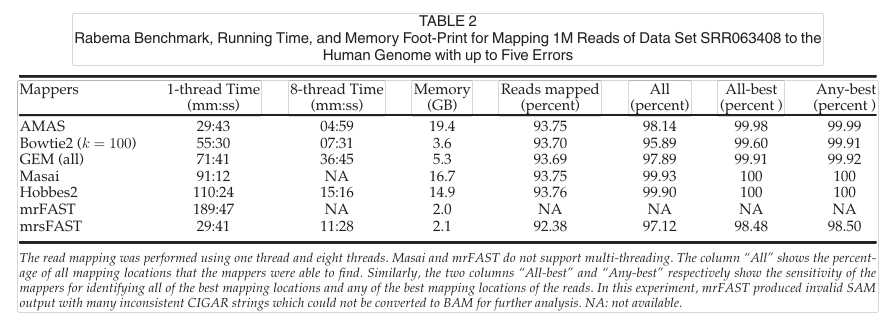
在处理大规模数据集上,AMAS在运行时间,映射速率和灵敏度方面优于GEM和mrsFAST。内存占用AMAS和GEM相比其他算法是比较高的,这也是自适应种子以后需要改进的地方。在八线程处理大规模数据集上,GEM有时会出现错误,内存占用量会显着增加甚至会崩溃。
AMAS通过参数“fse”控制最小种子数,如果设置过高将抑制自适应种子的优点,产生大量候选者并因此消耗更长的运行时间。设置”fse=2”时,AMAS花费的映射时间最少,所以将”fse=2”设为ASAM的默认值。
“F”为自适应种子的频率阈值,降低”F”会减少候选命中数,从而减少了映射时间。但然而,较低的“F”也导致种子长度过长,每个分区的种子过少,因此会降低映射灵敏度。而且减少“F”也扩大了自适应种子的索引树导致消耗了更多的内存。
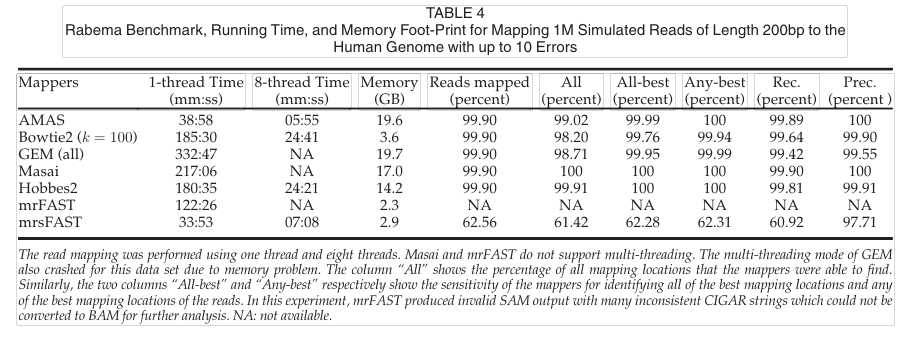
首先,AMAS在索引步骤中预先计算所有可能的自适应种子,以避免在映射步骤中重复不必要的自适应种子计算基于高度重复的最后种子和额外种子的精确过滤进一步大大收紧候选空间,有效地减少种子扩展所需的时间。并且AMAS允许用户控制每个读取分区的最小自适应种子数,从而在运行时间和灵敏度之间实现理想的平衡。在多线程环境中AMAS也可以很好地处理数据,并且无论数据的大小如何,都能保持内存占用稳定。
但是自适应种子的优势未得到充分发掘,目前GEM是唯一利用自适应种子的全映射器。这项技术仍需要继续研究。
标签:特点 数据集 display 额外 png 不同 效果 实现 内存占用
原文地址:https://www.cnblogs.com/liangfengqingye/p/9865330.html