标签:red selection rom 必须 append def oba ida weight
经常用到sklearn中的SVC函数,这里把文档中的参数:
本身这个函数也是基于libsvm实现的,所以在参数设置上有很多相似的地方。(PS: libsvm中的二次规划问题的解决算法是SMO)。
sklearn.svm.SVC(C=1.0,?kernel=‘rbf‘,?degree=3,?gamma=‘auto‘,?coef0=0.0,?shrinking=True,?probability=False,
Tol=0.001,?cache_size200,?class_weight=None,?verbose=False,? max_iter=-1,?
decision_function_shape=None,random_state=None)
? ?
参数:
1、C:C-SVC的惩罚参数C默认值是1.0,C越大,相当于惩罚松弛变量,希望松弛变量接近0,即对误分类的惩罚增大,趋向于对训练集全分对的情况,这样对训练集测试时准确率很高,但泛化能力弱。C值小,对误分类的惩罚减小,允许容错,将他们当成噪声点,泛化能力较强。
C一般可以选择为:0.0001 到10000,选择的越大,表示对错误例惩罚程度越大,可能会导致模型过拟合
2、kernel?:核函数,默认是rbf,可以是‘linear‘, ‘poly‘, ‘rbf‘, ‘sigmoid‘, ‘precomputed‘?
? 0 – 线性:u‘v
? 1 – 多项式:(gamma*u‘*v + coef0)^degree
? 2 – RBF函数:exp(-gamma|u-v|^2)
? 3 –sigmoid:tanh(gamma*u‘*v + coef0)
数学表达式:
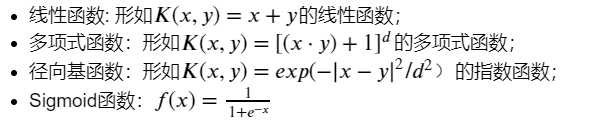
3、degree?:多项式poly函数的维度,默认是3,选择其他核函数时会被忽略。建议设置为2;
4、gamma?:‘rbf‘,‘poly‘和‘sigmoid‘的核系数。当前默认值为‘auto‘,它使用1 / n_features,如果gamma=‘scale‘传递,则使用1 /(n_features * X.std())作为gamma的值。当前默认的gamma‘‘auto‘将在版本0.22中更改为‘scale‘。
5、?coef0?:核函数的常数项。对于‘poly‘和 ‘sigmoid‘有用。
6、probability?:默认False。是否启用概率估计。必须在调用fit之前启用它,并且会减慢该方法的速度。
7、shrinking?:默认为true,是否采用shrinking heuristic(收缩启发式)方法
8、?tol?:默认为1e-3,停止训练的误差值大小,
9、cache_size?:默认为200,核函数cache缓存大小
10、?class_weight?:{dict,‘balanced‘}。将类i的参数C设置为SVC的class_weight [i] * C.?如果没有给出,所有类都应该有一个权重。"平衡"模式使用y的值自动调整与输入数据中的类频率成反比的权重n_samples?/?(n_classes?*?np.bincount(y))
11、verbose?:默认False。启用详细输出。请注意,此设置利用libsvm中的每进程运行时设置,如果启用,则可能无法在多线程上下文中正常运行。
12、max_iter?:最大迭代次数。-1为无限制。
13、decision_function_shape?:‘ovo‘, ‘ovr‘ or None, default=None3
14、random_state?:默认 无。伪随机数生成器的种子在对数据进行混洗以用于概率估计时使用。如果是int,则random_state是随机数生成器使用的种子;?如果是RandomState实例,则random_state是随机数生成器;?如果没有,随机数生成器所使用的RandomState实例np.random。
主要调节的参数有:C、kernel、degree、gamma、coef0。
? ?
与核函数相对应的libsvm参数建议:
1)对于线性核函数,没有专门需要设置的参数
2)对于多项式核函数,有三个参数。-d用来设置多项式核函数的最高此项次数,也就是公式中的d,默认值是3。-g用来设置核函数中的gamma参数设置,也就是公式中的第一个r(gamma),默认值是1/k(k是类别数)。-r用来设置核函数中的coef0,也就是公式中的第二个r,默认值是0。
3)对于RBF核函数,有一个参数。-g用来设置核函数中的gamma参数设置,也就是公式中的第一个r(gamma),默认值是1/k(k是类别数)。
4)对于sigmoid核函数,两个参数g以及r:gamma一般可选1 2 3 4,coef0选0.2 0.4 0.60.8 1
? ?
属性:
support_?:支持向量索引。
support_vectors_?:支持向量。
n_support_?:每一类的支持向量数目
dual_coef_?:决策函数中支持向量的系数
coef_?:赋予特征的权重(原始问题中的系数)。这仅适用于线性内核。
intercept_?:决策函数中的常量。
?
? ?
? ?
---实验练习
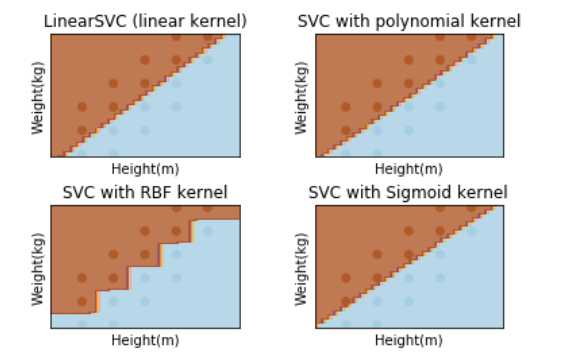
对身高体重的数据进行svm分类,要求分别使用线性函数、三次多项式函数、径向基函数和Sigmoid函数作为核函数进行模型训练,并观察分类效果(利用plt绘制样本点,胖的样本点与瘦的样本点用颜色区分,结果绘制出分类曲线):
? ?
代码:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 60 61 62 63 64 65 66 67 68 69 70 71 | %matplotlib inline import numpy as np import scipy as sp from sklearn import svm from sklearn.model_selection import train_test_split # 注意: 如果sklearn是0.18之前的版本,则执行下面的语句: # from sklearn.cross_validation import train_test_split import matplotlib.pyplot as plt #导入数据 data = [] labels = [] # 读取身高体重数据集 with open("./Input/data.txt") as ifile: for line in ifile: tokens = line.strip().split(‘ ‘) # data列表存储身高体重数据 data.append([float(tk) for tk in tokens[:-1]]) # label列表存储此人被定义为胖还是瘦 labels.append(tokens[-1]) # x: 以array形式存储身高体重构成的坐标 x = np.array(data) # labels: 将label列表array化 labels = np.array(labels) y = np.zeros(labels.shape) # y:存储0,1化的体重标签,值为0说明此人被定义为瘦,值为1说明此人定义为胖 y[labels==‘fat‘]=1 x_train, x_test, y_train, y_test = train_test_split(x, y, test_size = 0.0) # 建立画布(可以不用关心具体的实现过程) h = .02 x_min, x_max = x_train[:, 0].min() - 0.1, x_train[:, 0].max() + 0.1 y_min, y_max = x_train[:, 1].min() - 1, x_train[:, 1].max() + 1 xx, yy = np.meshgrid(np.arange(x_min, x_max, h), np.arange(y_min, y_max, h)) # 绘图名称 titles = [‘LinearSVC (linear kernel)‘, ‘SVC with polynomial kernel‘, ‘SVC with RBF kernel‘, ‘SVC with Sigmoid kernel‘] ? ? ‘‘‘ svm.SVC(kernel=‘linear‘):核函数为线性函数 svm.SVC(kernel=‘poly‘, degree=3):核函数为3次多项式函数,如果degree=n,则使用的核函数是n次多项式函数 svm.SVC():核函数为径向基函数 svm.SVC(kernel=‘sigmoid‘):核函数为Sigmoid函数 ‘‘‘ #以下是参数的重点部分: # 核函数为线性函数 clf_linear = svm.SVC(kernel=‘linear‘,C=50).fit(x, y) # 核函数为多项式函数 clf_poly = svm.SVC(kernel=‘poly‘, degree=2,coef0=10).fit(x, y) # 核函数为径向基函数 clf_rbf = svm.SVC(kernel=‘rbf‘,C=100,gamma=0.5).fit(x, y) # 核函数为Sigmoid函数 clf_sigmoid = svm.SVC(kernel=‘sigmoid‘,C=1000000,gamma=0.0001,coef0=-1.5).fit(x, y) ? ? for i, clf in enumerate((clf_linear, clf_poly, clf_rbf, clf_sigmoid)): answer = clf.predict(np.c_[xx.ravel(), yy.ravel()]) plt.subplot(2, 2, i + 1) plt.subplots_adjust(wspace=0.4, hspace=0.4) # 将数据点绘制在坐标图上 z = answer.reshape(xx.shape) plt.contourf(xx, yy, z, cmap=plt.cm.Paired, alpha=0.8) ? plt.scatter(x_train[:, 0], x_train[:, 1], c=y_train, cmap=plt.cm.Paired) plt.xlabel(‘Height(m)‘) plt.ylabel(‘Weight(kg)‘) plt.xlim(xx.min(), xx.max()) plt.ylim(yy.min(), yy.max()) plt.xticks(()) plt.yticks(()) plt.title(titles[i]) plt.show() |
? ?
实验结果:
? ?
? ?
? ?
? ?
? ?
? ?
? ?
? ?
? ?
? ?
? ?
? ?
? ?
? ?
标签:red selection rom 必须 append def oba ida weight
原文地址:https://www.cnblogs.com/yifanrensheng/p/11863324.html