标签:将不 min class color chain not ros this 简单
人类基因组有不同的版本,目前用的最多的是hg38和hg19,为了将不同版本的染色体上的位置一一对应,UCSC出了这款工具liftOver,官方的定义是:
This tool converts genome coordinates and genome annotation files between assemblies.
1 wget http://hgdownload.cse.ucsc.edu/admin/exe/linux.x86_64/liftOver
2.坐标注释文件下载
hg38Tohg19注释文件:
1 wget http://hgdownload.cse.ucsc.edu/goldenPath/hg38/liftOver/hg38ToHg19.over.chain.gz
hg19Tohg38注释文件:
1 wget http://hgdownload.cse.ucsc.edu/goldenPath/hg19/liftOver/hg19ToHg38.over.chain.gz
下载得到的*over.chain.gz不需要解压
如果需要其他版本的注释文件,请见Sequence and Annotation Downloads,下载对应的liftOver注释文件即可。
只接受BED格式文件,BED格式文件只定义前三列:chr start end,无表头
注:start不等于end(如果是单位点的话,建议所有end+1
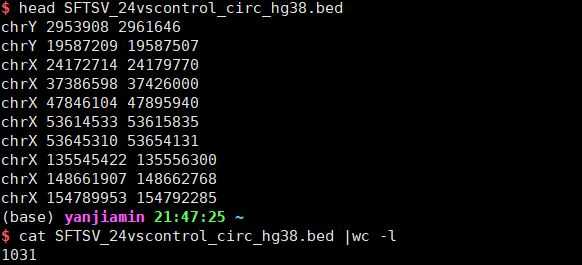
简单两个命令即可
1.将liftOver变为可执行文件
2.执行,参数为inputfile,over.chain.gz,outputfile,unmapfile(会输出没有对应上的行)
Example:
hg38>hg19
1 /path/to/liftOver /path/to/SFTSV_24vscontrol_circ_hg38.bed /path/to/hg38ToHg19.over.chain.gz /path/to/SFTSV_24vscontrol_circ_hg19.bed /path/to/SFTSV_24vscontrol_circ_hg19_unmap.bed
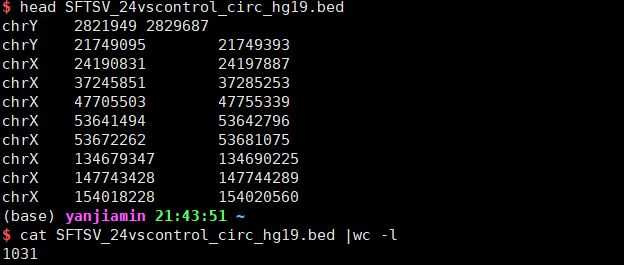
显示坐标已经完全转换
2019-12-10
22:03:03
标签:将不 min class color chain not ros this 简单
原文地址:https://www.cnblogs.com/yanjiamin/p/12019454.html