标签:archive tar panda normal 读取数据 gis 流程 image regress
逻辑回归,虽然有回归二字,但其实是个分类算法,主要用于二分类.
逻辑回归是吧线性回归得到的值,进行一个转换,来解决分类问题
输入范围-∞到+∞, 输出的值在[0,1]
公式是这样的
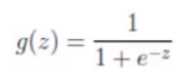
e为常数,如果z趋近于+∞,e的负z次就越接近于0,g(z)=1.如果z趋近于-∞,e的负z次就越来越大.分母越大,g(z)趋近于0.
曲线图:
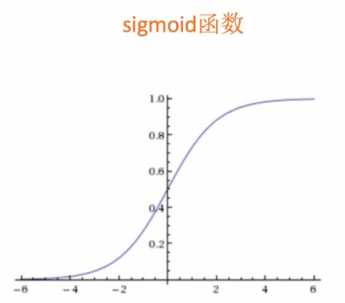
逻辑回归的公式:

逻辑回归的损失函数,优化(了解一下)

### 与信息熵有些相似,对数. ###
sklearn.linear_model.LogisticRegression(penalty=‘l2‘,C=1.0) Logistic 回归分类器 coef_:回归系数 自带L2正则化解决过拟合问题.(C参数.可修改)
项目背景:通过给定的良/恶性乳腺肿瘤数据,判断是否患癌症
数据:原始数据下载地址
数据描述:
1. 699条样本,共11列数据,第一列用于检索的id,后9列分别是与肿瘤相关的医学特征,最后一列表示肿瘤类型的数值
2. 包含16个缺失值,用‘?‘标出.
项目流程:
代码:
import pandas as pd import numpy as np from sklearn.model_selection import train_test_split #数据集划分 from sklearn.preprocessing import StandardScaler #数据标准化 from sklearn.linear_model import LogisticRegression #逻辑回归 from sklearn.metrics import classification_report # 召回率...deng # 读取数据 提前构造列名 columns = [‘Sample code number‘,‘Clump Thickness‘,‘Uniformity of Cell Size‘,‘Uniformity of Cell Shape‘,‘Marginal Adhesion‘,‘Single Epithelial Cell Size‘,‘Bare Nuclei‘,‘Bland Chromatin‘,‘Normal Nucleoli‘,‘Mitoses‘,‘Class‘] data = pd.read_csv(‘breast-cancer-wisconsin.data‘,names=columns) # data.head() # 数据处理-缺失值 data = data.replace(to_replace=‘?‘,value=np.NaN) # 删除 有缺失值的不要 data = data.dropna() # 分割数据集 x_train, x_test,y_train,y_test =train_test_split(data[columns[1:10]],data[columns[10]],test_size=0.25) # 1参数为特征,2参数目标值 # ↑除去id不要 ,其他的列都要 # 标准化 std_x = StandardScaler() x_train = std_x.fit_transform(x_train) x_test = std_x.transform(x_test) # 目标值需要做标准化吗? 不需要,分类 # 逻辑回归预测 lg = LogisticRegression() lg.fit(x_train,y_train) print(‘准确率:‘,lg.score(x_test,y_test)) #不是我们需要的,知道也没用,要的是召回率 y_predict = lg.predict(x_test) print(‘召回率:‘,classification_report(y_test,y_predict,labels=[2,4],target_names=[‘良性‘,‘恶性‘])) # classification_report参数:1. 真实值,2. 预测值,3. labels 分类内的值,4. target_names 对分类结果命名 # 后续还可以做交叉验证,或调整一下参数
输出结果:
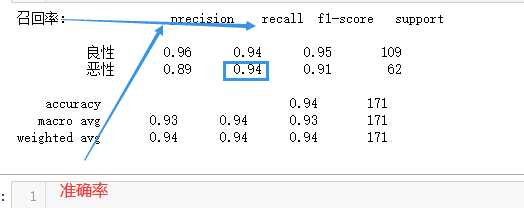
### 0.94,100个有6个没检查出来,放跑了,运气好. ###
标签:archive tar panda normal 读取数据 gis 流程 image regress
原文地址:https://www.cnblogs.com/luowei93/p/12635194.html