标签:输入数据 变换 col efi -o 参数设置 处理 none 表达
PIE-Hyp参量反演
高光谱数据波谱分辨率高,可精确识别地物波谱特征,捕捉同种地物间微小差别,为地物的定量反演提供帮助,因而高光谱应用范围极其广泛。
在地质学领域中,矿物中金属离子Fe3+、Fe2+、Mn2+的电子跃迁在可见光、近红外光谱区域形成典型的光谱波形,矿物中官能团OH-、CO32-及Si-O键等的振动在短波红外光谱区域形成一系列的吸收特征,这些诊断性的吸收特征构成成像光谱识别矿物的理论基础。在土壤有机质含量、离子含量、湿度及土壤侵蚀与退化等土壤研究方面,高光谱遥感数据提供了连续窄波段的短波红外光谱信息,使得土壤评价与监测有了更强有力的工具。
PIE-Hyp定量反演模块中基于连续统去除算法和一阶微分算法处理反射率数据,利用多元逐步线性回归法建立物质的定量反演模型供用户选择。PIE-Hyp定量反演模块中模型属于线性模型,高光谱定量反演模块需用户自己建立线性模型来提供相应的模型参数,应准确对应建模时的波段所对应的波长。
通过PIE-Hyp对原始影像进行参量反演。
序号 | 数据名称 | 数据说明 |
1 | Cup99hy.tiff | 原始影像 |
2 | CupriteRefiectance.dat | 叶绿素a浓度计算数据文件 |
在"定量应用"标签下的"参量反演"组,单击【定量反演】按钮,弹出"定量反演"对话框,如下图所示:
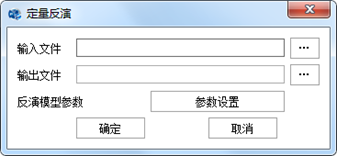
图 31 定量反演对话框
点击【…】按钮,弹出输入数据信息对话框;
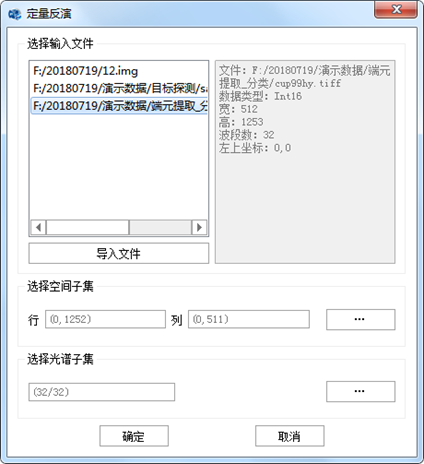
图 32 定量反演输入数据对话框
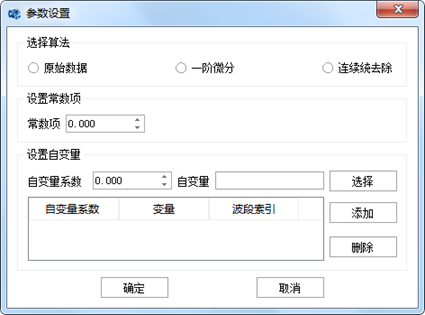
图 33 参数设置对话框
Fe3+=3.001-142.147*b38(732.07nm )+61.473*b44(793.13 nm)-203.345*b25
(599.80 nm)+281.072*b28(630.32 nm)-2.8*b215(2304.71 nm)-100.675*b24
(589.62 nm)
Fe3+=2.543-2498.9*b15(498.04nm)+1933.03*b114(1285.76nm)-6529.17*b151
( 1659.000nm)+7321.59*b152(1669.100nm)-1740*b134 (1487.530nm)
Fe3+=-9.171-53.797*b19(538.74nm)-165.898*b97(1114.19nm)+25.284*b47(823.65nm)-29.188*b53(884.704nm)+51.085*b92(1063.79nm)+120.481*b99(1134.38)+105.3*b15(498.04nm)-46.792*b13(477.69)+15.413*b205(2203.83nm)
(1)原始光谱模型表达式:R2=0.863,adjusted R2=0.845,RMSE=0.159
SOM=3.448+2.029*b41(762.600nm )+71.088*b52(874.530nm)+7.396*b53(884.704nm)-95.985*b54(894.880nm)-22.185*b85(1027.16nm)+28.911*b93(1073.889nm)
(2)连续统去除模型表达式:R2=0.883,adjusted R2=0.874,RMSE=0.163
SOM=8.79-4.341*b40(752.425nm)+13.231*b52(874.530nm)-16.870*b54(894.880nm)
所有参数设置完成后,点击定量反演对话框上的【确定】按钮,进行反演操作。
暂无。
问题1:颜色指数反演对输入的高光谱影像有何要求?
解决办法:输入影像应为经过大气校正的反射率数据。
问题2:营养状态指数反演对输入的数据有何要求?
解决办法:将反演叶绿素a浓度功能的输出结果作为该项的输入。
(1) 使用课程提供的课后练习数据,实践参数反演过程。
标签:输入数据 变换 col efi -o 参数设置 处理 none 表达
原文地址:https://www.cnblogs.com/PIESat/p/14068018.html