标签:
为了验证杂交的质量,Affymetrix公司加入了两类嵌入探针组:
一、poly-A内参:包括lys、phe、thr、dap
对应的探针组名称为:AFFX-r2-Bs-lys-3_at、AFFX-r2-Bs-dap-3_at、
AFFX-r2-Bs-phe-3_at、AFFX-r2-Bs-thr-3_s_at
(注意:一般只取3’的表达值)
期望检测到的信号强度为lys<phe<thr<dap
期望lys检测值(Detection Call)为P
以样品GSM362947_LTT.CEL和GSM362948_LTT.CEL为例:
1)获取lys、phe、thr、dap的信号强度
library(yaqcaffy)
rawData<-ReadAffy("GSM362947_LTT.CEL","GSM362948_LTT.CEL")
yaqcResult<-yaqc(rawData)
> yaqcResult@morespikes[grep("(lys|phe|thr|dap).*3",rownames(yaqcResult@morespikes), ignore.case = TRUE),]
GSM362947_LTT.CEL GSM362948_LTT.CEL
dap3 621.85881 618.93019
thr3 157.46776 141.04182
lys3 26.22679 24.34060
phe3 80.33482 78.98733
在这个例子中,两个样品的lys、phe、thr、dap信号强度都符合lys<phe<thr<dap
2)arrayanalysis的问题
用R语言软件arrayanalysis分析以上样品得出的结论却是:
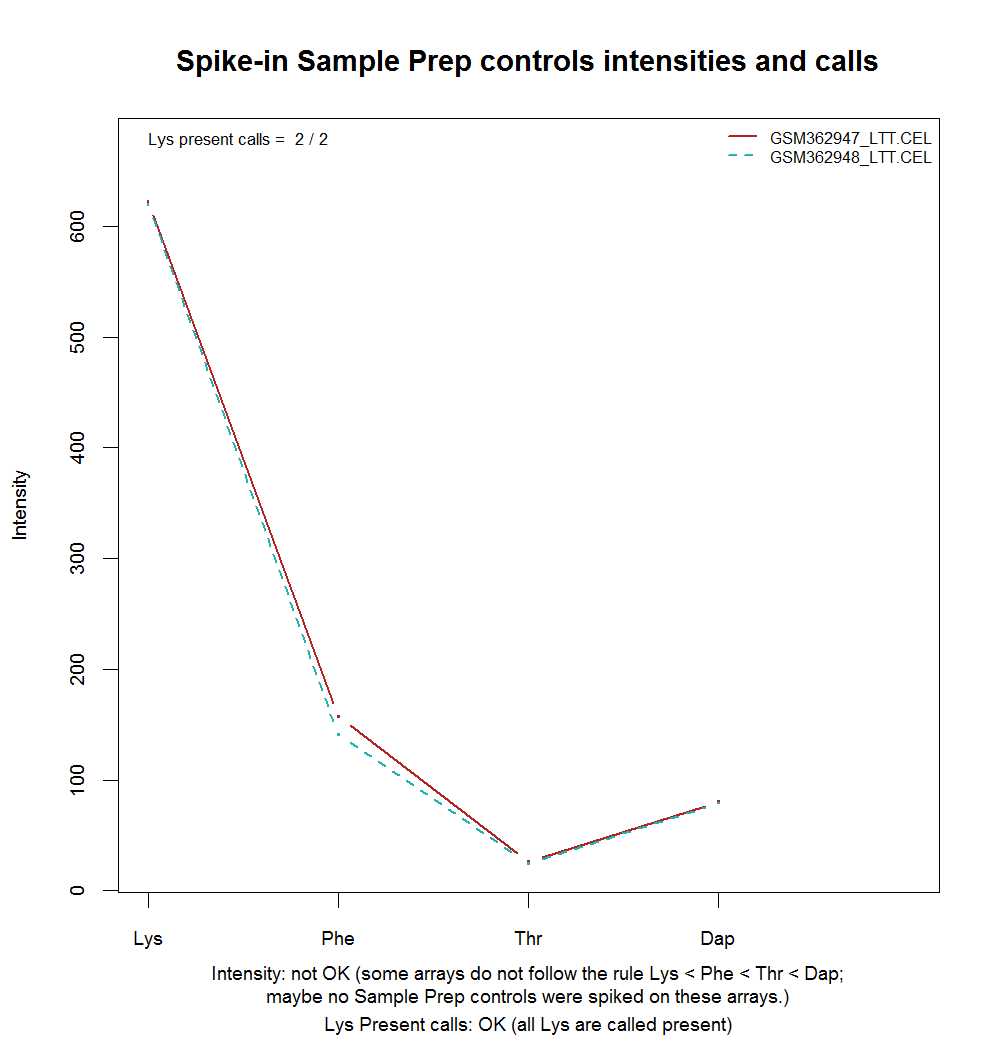
这明显不符合lys<phe<thr<dap,这是为什么?
我们先来看看yaqcResult@morespikes是什么:
GSM362947_LTT.CEL GSM362948_LTT.CEL
b5 102.92636 123.20557
b3 98.75225 106.84035
bm 113.12133 129.45077
c5 298.87382 322.52600
c3 361.89032 421.07440
d5 1289.20872 1334.69642
d3 1489.85897 1702.89382
dap5 303.57364 156.58720
dap3 621.85881 618.93019
dapm 414.50848 333.24881
thr5 108.41316 39.60845
thr3 157.46776 141.04182
thrm 129.56930 98.51053
lys5 23.28581 21.26793
lys3 26.22679 24.34060
lysm 30.29455 28.88963
phe5 31.29219 12.49173
phe3 80.33482 78.98733
phem 47.31779 32.76261
在arrayanalysis的源码functions_imagesQC.R中,先用正则表达提取出yaqcResult@morespikes中符合"(lys|phe|thr|dap).*3"条件的:
> spnames<-rownames(yaqcResult@morespikes[grep("(lys|phe|thr|dap).*3", # only 3‘ !
+ rownames(yaqcResult@morespikes), ignore.case = TRUE),])
> spnames
[1] "dap3" "thr3" "lys3" "phe3"
注意到spnames的顺序是"dap3" "thr3" "lys3" "phe3"而不是"lys3" "phe3" "thr3" "dap3"
然后把名称为spnames的探针组也就是"dap3" "thr3" "lys3" "phe3"的信号强度赋给sprep
> sprep<-t(yaqcResult@morespikes[spnames,])
> sprep
dap3 thr3 lys3 phe3
GSM362947_LTT.CEL 621.8588 157.4678 26.22679 80.33482
GSM362948_LTT.CEL 618.9302 141.0418 24.34060 78.98733
sprep的数据就直接用来作图了,所以上图的信号强度趋势的顺序应该是dap、thr、lys、phe,而不是预想的lys、phe、thr、dap
(现在我所使用的arrayanalysis版本确实存在这个问题,但是也许以后会改进)
3)获取lys的检测值:
calls<-detection.p.val(rawData)$call
lysCall<-calls[rownames(calls)[grep("AFFX-r2-Bs-lys-3_at",rownames(calls), ignore.case = TRUE)],]
> lysCall
GSM362947_LTT.CEL.present GSM362948_LTT.CEL.present
"P" "P"
结论:两个样品的lys的检测值都为P
二、杂交内参:包括BioB、BioC、BioD、CreX
对应的探针组名称为:AFFX-r2-Ec-bioB-3_at、AFFX-r2-Ec-bioC-3_at、
AFFX-r2-Ec-bioD-3_at、AFFX-r2-P1-cre-3_at
(注意:一般只取3’的表达值)
期望检测到的信号强度为BioB<BioC<BioD<CreX
期望BioB检测值(Detection Call)为P
以样品GSM286756.CEL为例:
1)获取BioB、BioC、BioD、CreX的信号强度
library(simpleaffy)
qcResult<-qc(rawData)
> spikeInProbes(qcResult)
AFFX-r2-Ec-bioB-3_at AFFX-r2-Ec-bioC-3_at
GSM362947_LTT.CEL 6.625742 8.499409
GSM362948_LTT.CEL 6.739313 8.717931
AFFX-r2-Ec-bioD-3_at AFFX-r2-P1-cre-3_at
GSM362947_LTT.CEL 10.54096 12.37574
GSM362948_LTT.CEL 10.73377 12.48219
在这个例子中,两个样品的BioB、BioC、BioD、CreX信号强度都符合BioB<BioC<BioD<CreX
2)获取BioB的检测值:
> show(yaqcResult)
GSM362947_LTT.CEL GSM362948_LTT.CEL
……
AFFX-r2-Ec-bioB-3_at_call "P" "P"
……
结论:两个样品的BioB检测值都为P
三、计算poly-A内参和杂交内参的信号强度表达值采用了JustMAS算法,芯片中必须含有MM探针。除此之外,芯片中还要包含AFFX-r2-Bs-lys-3_at、AFFX-r2-Bs-dap-3_at、AFFX-r2-Bs-phe-3_at、AFFX-r2-Bs-thr-3_s_at、AFFX-r2-Ec-bioB-3_at、AFFX-r2-Ec-bioC-3_at、AFFX-r2-Ec-bioD-3_at、AFFX-r2-P1-cre-3_at探针组。不过这些步骤都由yaqc和qc函数完成了,读者不必过多关心JustMAS算法。yaqc和qc有一点不同,就是计算出信号强度表达值x后,yaqc会取2的x次方,qc则是2的x次方再取2的对数,也就是又变回x了。
07、poly-A内参和杂交内参(arrayanalysis的问题)
标签:
原文地址:http://www.cnblogs.com/xianwen/p/4522286.html